ここまで、タンパク質の合成につながるRNAポリメラーゼIIによる転写を中心に説明してきた。ここでは、rRNAの合成に関わるRNAポリメラーゼIと、tRNAや5S rRNAの合成に関わるRNAポリメラーゼIIIによる転写についても紹介したい。ついでに、rRNA前駆体の転写からリボソームの組み立てまでのプロセスも紹介したいと思う。
真核生物の3種類のRNAポリメラーゼと基本転写因子
真核生物のRNAポリメラーゼの頁ですでに説明したが、真核生物にはRNAポリメラーゼI〜IIIの3種類のRNAポリメラーゼが存在する。これらの3種類のRNAポリメラーゼは転写の対象となる遺伝子群が異なっており、RNAポリメラーゼIはrRNA前駆体、RNAポリメラーゼIIはmRNA前駆体、RNAポリメラーゼIIIは5S rRNA、tRNAなどの小分子RNA遺伝子を転写する。
また、真核生物のRNAポリメラーゼは単独では転写を開始できないため、転写開始には基本転写因子を必要とする。これはRNAポリメラーIIに限らず、RNAポリメラーゼIやIIIにも共通した特徴である。
RNAポリメラーゼIによる転写
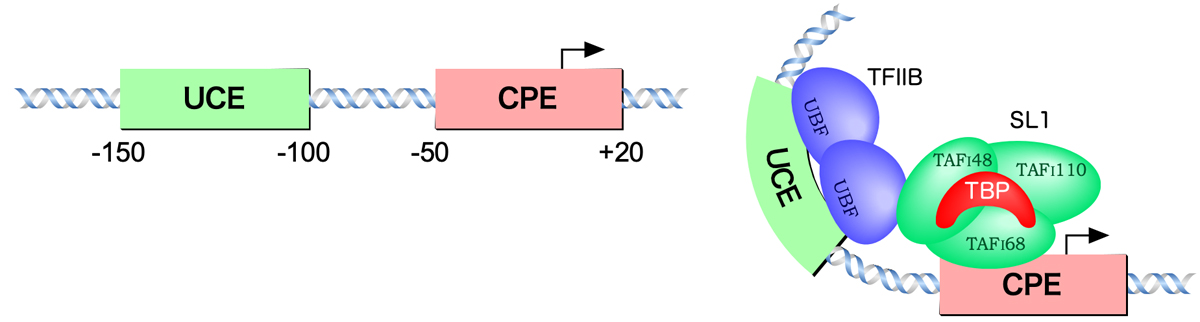
RNAポリメラーゼIは、rRNA前駆体の転写を行う。真核生物の成熟rRNAのうち、28S, 18S, 5.8S RNAの3つは、このrRNA前駆体から作られる。rRNA遺伝子のプロモーターは、−200〜+20の範囲にあり、そこには2つのコンセンサス配列が存在する。位置は生物種によって多少異なるが、−150〜−100の範囲に上流制御エレメント(UCE:Upstream Control Element)、−50〜+10の範囲にコアプロモーターエレメント(CPE:Core Promoter Element)が存在する。そして、UCEに結合する基本転写因子はUBF(Upstream Binding Factor)であり、CPEに結合するのがSL1(Selective Factor 1)である。
SL1は、マウスとヒト間で抽出液(SL1)とプロモーターの組み合わせを変えるとin vitroで転写が生じないことから、抽出液とプロモーターの組み合わせの選択性に関わる因子ということで、このように命名された。SL1は、ヒトではTAFI48 (TAF1A), TAFI63 (TAF1B), TAFI110 (TAF1C), TBPの4つのサブユニットで構成される。なんと、SL1にはTBPが含まれるのである。TAFI68を介してCPEに結合し、TAFI48を介してUBFと相互作用する。このSL1の結合が、RNAポリメラーゼIをプロモーターに呼び込み、正確な転写開始点を決定している。
UBFは二量体を形成してUCEに結合する。UBFの結合によりDNAは大きく曲がり、これによってCPEとSL1の結合が安定化するらしい。
RNAポリメラーゼIIIによる転写
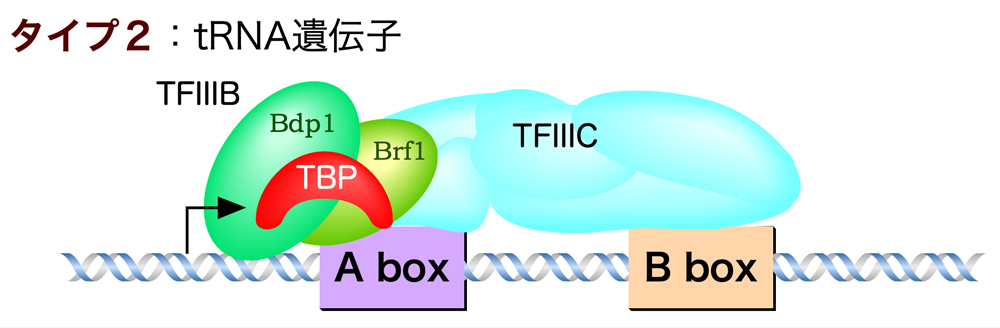
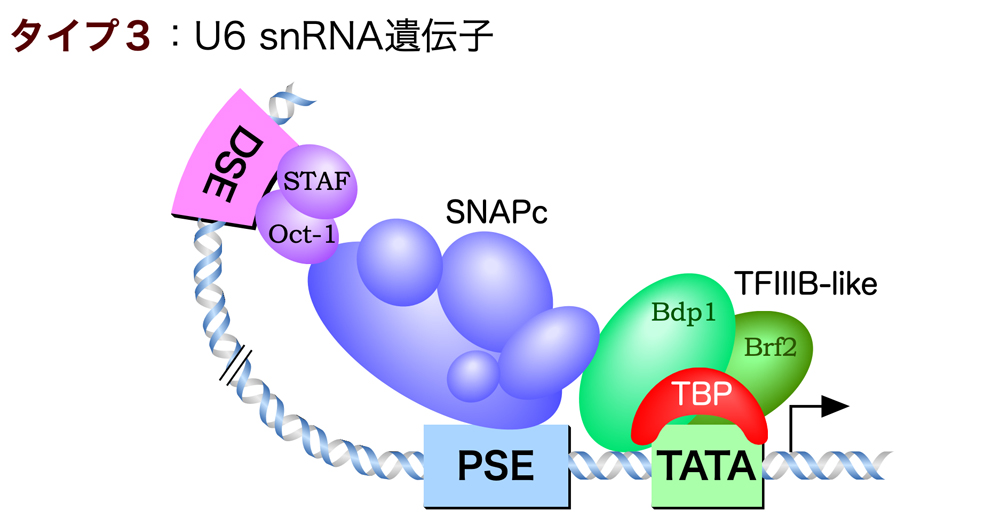
RNAポリメラーゼIIIは、tRNAや5S RNAなどの低分子RNAの転写を行う。rRNAの中でも、5S rRNAだけはRNAポリメラーゼIIIによって転写されるのである。RNAポリメラーゼIIIによって転写されるプロモーターは、その構造によりタイプ1〜3に分類される。その中でもタイプ1とタイプ2は、転写開始点よりも下流(遺伝子の内部)にプロモーターをもつ。
タイプ1のプロモーターは、tRNA遺伝子にみられる。転写開始点より下流にICR(Internal Control Region)があり、その中にBox AとIE(Internal Element)とBox Cが存在する。はじめにICRに基本転写因子TFIIIAが結合し、これによりICR全体をおおうようにTFIIICが結合する。TFIIICによりRNAポリメラーゼIIIの呼び込みに必要なTFIIIBがリクルートされ、転写開始点からその上流あたりに結合する。TFIIIBにはTBPが含まれるが、このときDNAへの結合を担うのはBrf-1である。
タイプ2のプロモーターは、5S rRNA遺伝子にみられる。転写開始点より下流にBox AとBox Bが存在し、そこに基本転写因子TFIIICが結合する。そして、TFIIICによりTFIIIBがリクルートされ、RNAポリメラーゼIIIが呼び込まれる。
タイプ3のプロモーターはU6 snRNA遺伝子にみられ、RNAポリメラーゼIIのプロモーターと同様に転写開始点より上流にプロモーターが存在する。しかも、TATAボックスまで存在する。また、TATAボックスの近くにはPSE(Proximal Sequence Element)があり、ここには基本転写因子SNAPcが結合する。そして、TATAボックスにはTFIIIB-likeが結合する。TFIIIB-likeではBrf1に代わってBrf2が使われており、このときTATAボックスに結合するのはTBPである。さらに上流には、コアプロモーターからの転写を高めるDSE(Distal Sequence Element)があり、ここには転写因子のOct-1やSTAFが結合する。
rRNA前駆体のプロセシング
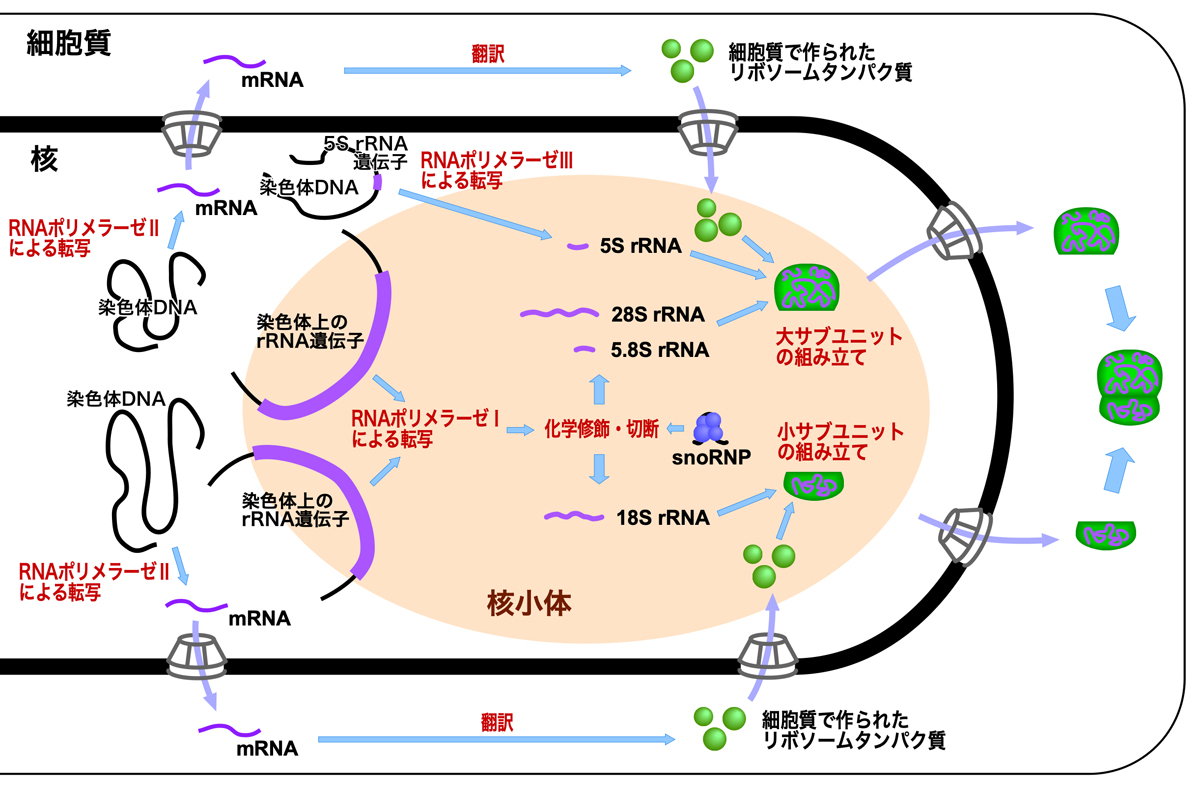
28S, 18S, 5.8S RNAの3つは、45S rRNA前駆体としてRNAポリメラーゼIにより転写される。この前駆体から成熟rRNAができるまでの過程を説明しよう。45S rRNA前駆体は、はじめに2種類の化学修飾を受ける。1つは、ウリジンの異性化によるシュードウリジン(プソイドウリジンともいう)の形成であり、もうひとつの修飾が、2’-O-メチル化による2’-O-メチルリボースの形成である。ヒトの場合、どちらの修飾もrRNA前駆体中の約100箇所で行なわれるが、その修飾部位を決めているのはsnoRNA(核小体低分子RNA)である(rRNAの化学修飾とsnoRNAのはたらきについては、別頁で詳細に説明することにする)。
最後にrRNA前駆体が切断されて、成熟した28S, 18S, 5.8S rRNAが完成する。そして、28S rRNAと5.8S rRNAに5S rRNAとリボソームタンパク質を合わせてリボソームの大サブユニットが、18S rRNAとリボソームタンパク質を合わせてリボソームの小サブユニットが形成される。
核小体におけるリボソームの組み立て
リボソームはタンパク質合成(翻訳)の場であり、哺乳類の細胞中には1000万個のリボソームがあると言われる。そして、rRNAはリボソームの構成成分であるため、ひとつの細胞には1000万分子のrRNAが必要になる。とにかく大量のrRNAが必要であり、細胞がもつ全RNAの約80%がrRNAである。
大量のrRNAを合成するために、ヒトのゲノム1セットには約200個のrRNA遺伝子があり、5本の染色体に分散して存在している。真核細胞の間期の核では、5本の染色体に分散したrRNA遺伝子が、核小体に集まる。核小体は、円滑にリボソームの組み立てを行うためのリボソーム製造装置である。
核小体の中でRNAポリメラーゼIによってrRNA遺伝子が転写され、snoRNAを介した修飾と切断により、28S, 18S, 5.8S rRNAが作られる。また、核質ではRNAポリメラーゼIIIにより5S rRNAが転写される。さらに、RNAポリメラーゼIIによりリボソームタンパク質遺伝子が転写され、そのmRNAは細胞質で翻訳され、合成されたリボソームタンパク質が核小体へと輸送される。
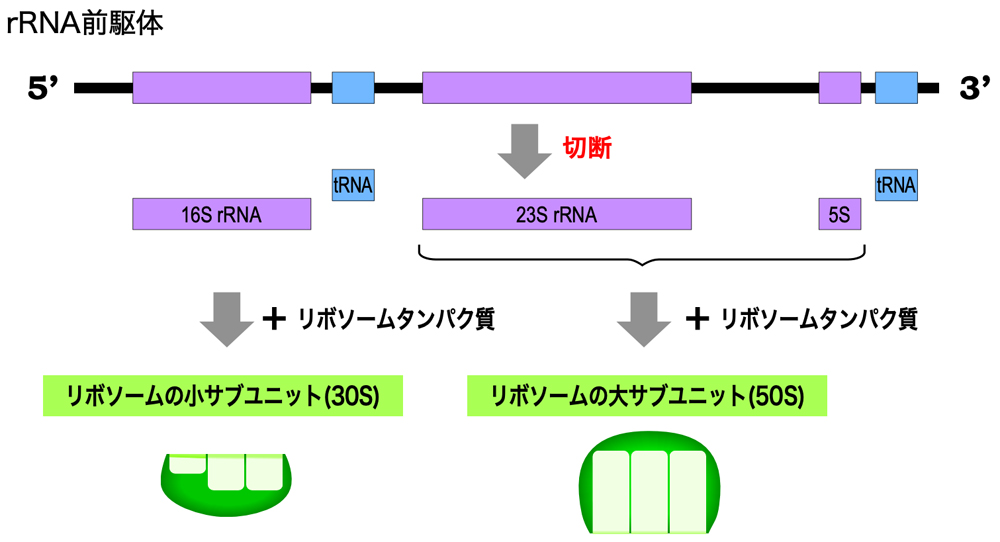
ちなみに、細菌(大腸菌)のリボソームを構成する16S, 23S, 5S rRNAも、rRNA前駆体として転写され、その後加工される。大腸菌は、ゲノム中に7つのrrnオペロンをもつことにより、大量のrRNA合成を可能にしている。また、大腸菌のrRNAにもシュードウリジンや2’-O-メチルリボースといった修飾が少数あるが、これらはタンパク質(修飾酵素)によって修飾される。