さて、いよいよ翻訳です。塩基配列としての遺伝情報が、アミノ酸配列として酵素活性などの活動に変換される重要なプロセスです。まずは、ポリペプチド鎖を伸長していくプロセスを紹介しましょう。
ポリペプチド鎖の伸長
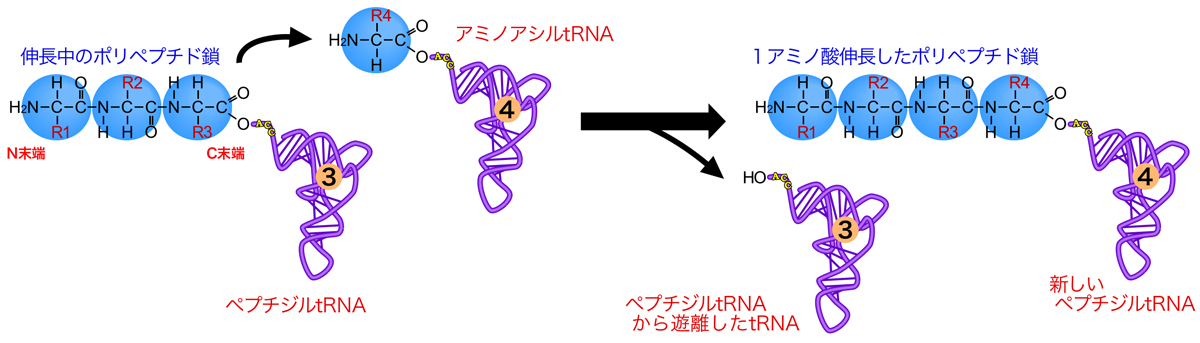
翻訳は、mRNA上の塩基配列にしたがって、mRNAの5’側からコドンを1つずつアミノ酸に変換していく過程である。そして、ポリペプチド鎖のC末端に1個ずつアミノ酸を付加していく。その伸長の基本プロセスは、以下の通りである。
まず、伸長中のプリペプチド鎖がペプチジルtRNAとして存在する。そしてその隣に、次に付加されるべきアミノ酸を運ぶアミノアシルtRNAがやってくる。すると、ペプチジルtRNAのポリペプチド鎖とtRNAをつなぐ高エネルギー結合が切断されて切り離される。そのエネルギーを利用して、ポリペプチド鎖のC末端と隣のアミノアシルtRNAに結合したアミノ酸のアミノ基(NH2基)との間でペプチド結合が形成される。結果として、1アミノ酸分伸長した新しいペプチジルtRNAが形成されるのである。
リボソームの構成
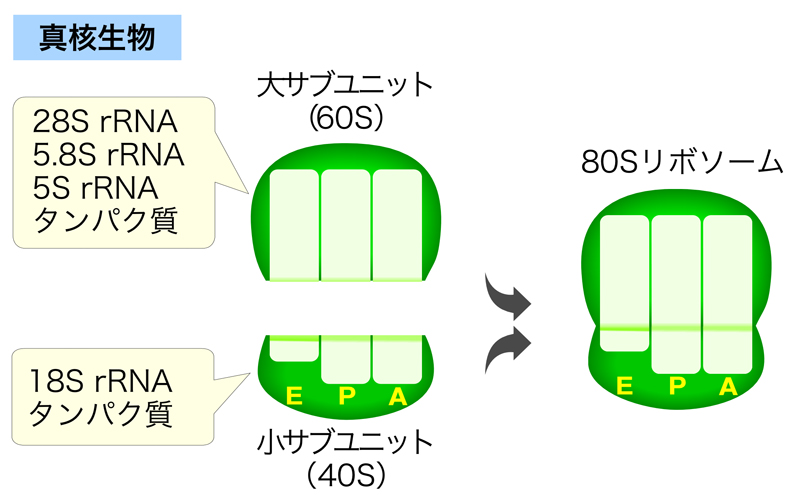
ポリペプチド鎖の伸長は、細胞質にあるリボソームで行われる。リボソームは、50種類以上のタンパク質と数種類のrRNAからなる複合体で、その構成は原核生物と真核生物でよく似ている。
リボソームは、小サブユニットと大サブユニットの2つで構成される。小サブユニットはtRNAのアンチコドンとmRNAのコドンを正確に対応させる役割をもち、大サブユニットはアミノ酸間にペプチド結合を形成する役割をもつ。
リボソームの主要な構成成分はrRNAであり、原核生物の大サブユニット(50S)を構成するのは23Sと5S rRNA、原核生物の小サブユニット(30S)を構成するのは16S rRNAである。一方、真核生物では、大サブユニット(60S)を構成するのは28Sと5.8Sと5S rRNA、小サブユニット(40S)を構成するのは18S rRNAである。
リボソームには、機能的に重要な以下の3つの部位が存在する。
- A部位:アミノアシルtRNAの結合部位
- P部位:ペプチジルtRNAの結合部位
- E部位:tRNAの出口
リボソーム上でのポリペプチド鎖伸長
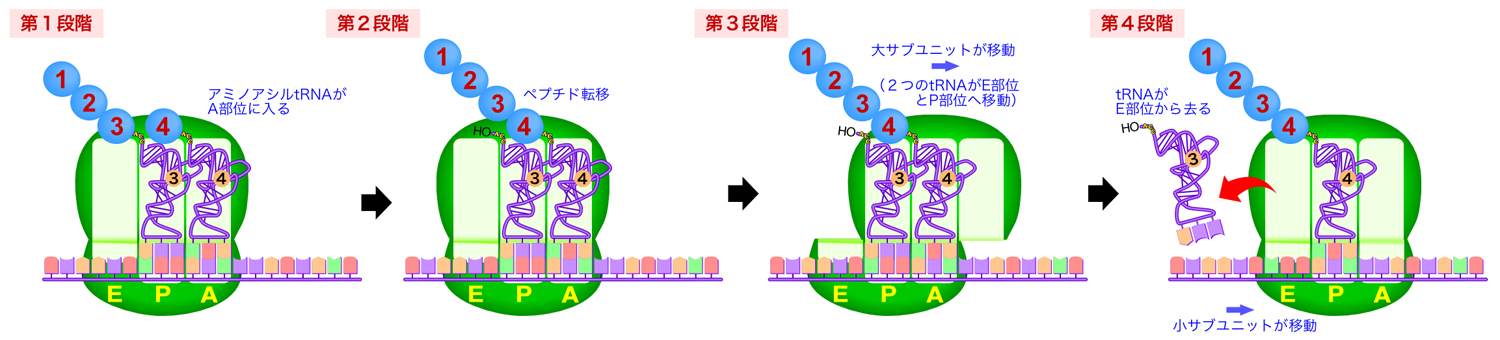
リボソーム上でのポリペプチドさの伸長は、以下の4段階の反応で行われる。ここ(下の図)では、前のサイクルが終了し、E部位にtRNA、P部位にペプチジルtRNAがいるところからスタートする。
- 空いているA部位に次に付加されるべきアミノ酸を運ぶアミノアシルtRNAが入る。
- ポリペプチド鎖のC末端がP部位のtRNAから外れ、A部位のtRNAに結合したアミノ酸のアミノ基(NH2基)と新しいペプチド結合を形成する(ペプチド転移)。
- 小サブユニットが保持するmRNAに対して大サブユニットが移動する。その結果、P部位とA部位にいる2つのtRNAの受容ステム部分が、それぞれ大サブユニットのE部位とP部位へ移動することになる。
- tRNAのアンチコドン部分とmRNAに対して、小サブユニットが3ヌクレオチド分移動する。役目を終えたE部位にいるtRNAは、リボソームを去る。
伸長因子
上述のような基本プロセスで翻訳は進行するが、ここではもう少し詳しく説明しよう。
リボソームでの翻訳では、伸長因子(elongation factor)が関与する。伸長因子がなくてもタンパク質を合成することはできるが、その場合非常に遅く不正確になるらしい。そこで、翻訳を効率よく高い精度で行うために、伸長因子が必要となる。大腸菌を例に紹介すると、以下の3つの伸長因子が関与する。
- EF-Tu
グアノシン三リン酸(GTP)と結合してEF-Tu•GTPとして作用し、アミノアシルtRNAに結合して翻訳の進行を助けるとともに、翻訳の精度を高める役割をもつ。すべてのtRNAは、EF-Tuと結合しているようである。 - EF-G
グアノシン三リン酸(GTP)と結合してEF-G•GTPとして作用し、tRNAの移動を促す役割をもつ。 - EF-Ts
GTPが加水分解されてグアノシン二リン酸(GDP)となったEF-Tu•GDPを、EF-Tu•GTPに変換する。
では、順を追って説明しよう。
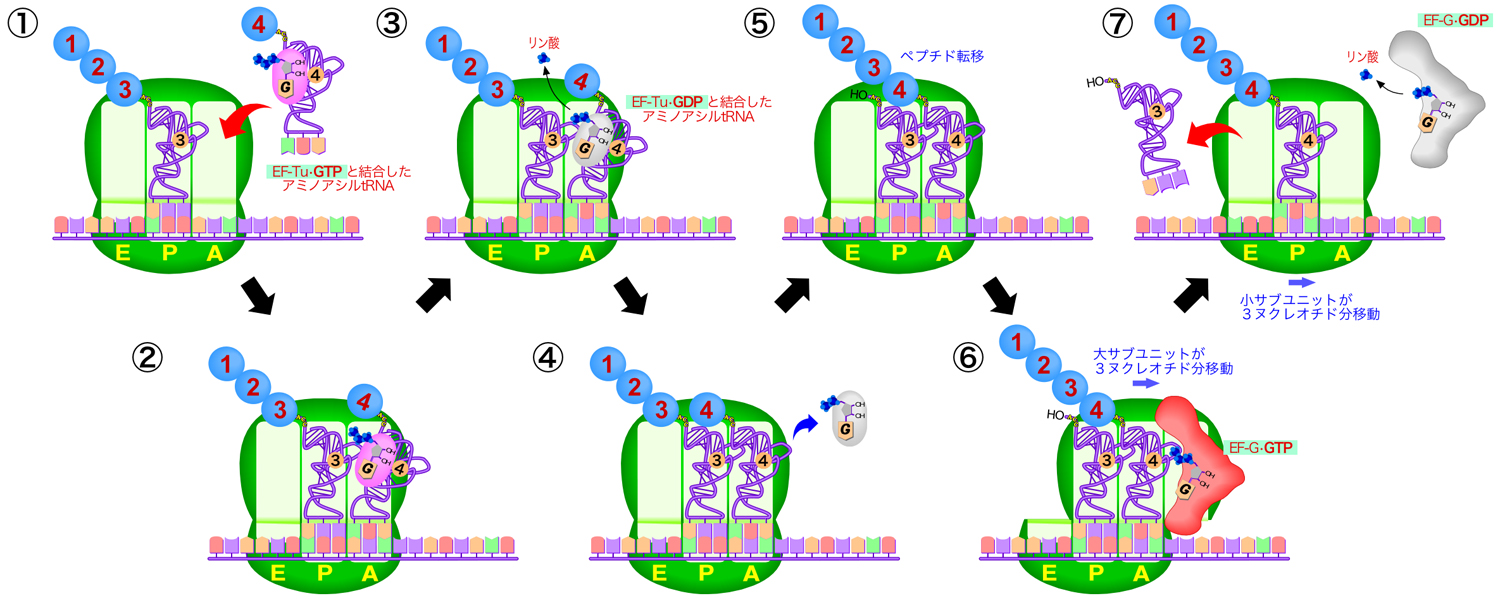
- 空いているA部位に、次に付加されるべきアミノ酸を運ぶアミノアシルtRNAがEF-Tu•GTPを伴ってやってくる。
- 小サブユニット側ではアミノアシルtRNAがA部位に入り、コドン-アンチコドンを対合させる。しかし大サブユニット側では、EF-Tu•GTPによってA部位への進入が妨げられ、アミノアシルtRNAはA部位に入れない状態になる。この間にコドン-アンチコドンが正しく対合しているか確認され、正しくない場合、このアミノアシルtRNAはリボソームから外れてしまう。
- 小サブユニット側でコドン-アンチコドンの対合が正しいことが確認されると、EF-Tuに結合したGTPがGDPへと加水分解される。
- EF-Tu•GDPはアミノアシルtRNAから離れ、大サブユニット側でもアミノアシルtRNAはA部位に入り、タンパク質合成に利用できるようになる。アミノアシルtRNAがEF-Tu•GDPから解放されてA部位に入るまでのわずかな時間でも、コドン-アンチコドンの対合が正しくない場合は、アミノアシルtRNAはリボソームから外れることができる。こうして、翻訳の精度を99.99%まで高めている。
- ポリペプチド鎖のC末端がP部位のtRNAから外れ、A部位のtRNAに結合したアミノ酸と新しいペプチド結合を形成する(ペプチド転移)。
- ペプチド転移が起こると、大サブユニットが移動(回転)してずれることにより、P部位とA部位にいる2つのtRNAの受容ステム部分が、それぞれ大サブユニットのE部位とP部位へ移動することになる。EF-G•GTPは、この状態を安定化する。
- EF-Gに結合したGTPがGDPへと加水分解されると、EF-G•GDPはリボソームから解離し、小サブユニットが3ヌクレオチド分移動(回転)することで、もとの状態に戻る。
このような伸長因子を作用は、真核生物でも極めて類似しているようで、真核生物にはEF-Tuに該当するeEF1A(EF-1α)、EF-Tsに該当するeEF1B(EF-1β)、EF-Gに該当するeEF2(EF-2)が存在する。
翻訳の正確性
遺伝暗号は、非常に正確に翻訳される。その仕組みのひとつは、アミノアシルtRNA合成酵素が、正確にtRNAとアミノ酸とを結びつける点にある。また正しい組み合わせでtRNAとアミノ酸が結合していると、EF-Tuとの親和性も高くなり、結果としてEF-Tuが正しいアミノアシルtRNAを選んでリボソームへ運ぶことになる。
さらに、小サブユニットのrRNAがコドン-アンチコドン塩基対といくつかの水素結合を作り、組み合わせの正しさを判断しているようである。下の図は、mRNA中のUUUコドン(フェニルアラニンをコード)とtRNAのGAAアンチコドンを示している。コドンの3番目はゆらぎの位置に該当するため、この相互作用はそんなに厳密ではないようである。
翻訳の速度
余談だが、翻訳の速度ってどのくらいだと思いますか?
実はリボソームにおける翻訳の速度は、真核生物で1秒間に2アミノ酸、原核生物では1秒間に20アミノ酸である。上述のように、コドン-アンチコドンの組み合わせを確かめて、EF-Tuに結合したGTPを加水分解して、ペプチド転移をして...と悠長にやっていては、とてもそんな速度ではできない。細胞の中では、我々のイメージをはるかに超える速度で、さまざまな仕組みが働いているようである。