1985年、George P. Smith博士により繊維状ファージをもちいたファージディスプレイ法が開発されました。そこで、2018年のノーベル化学賞に関連して、「ファージディスプレイ法」を紹介しましょう。
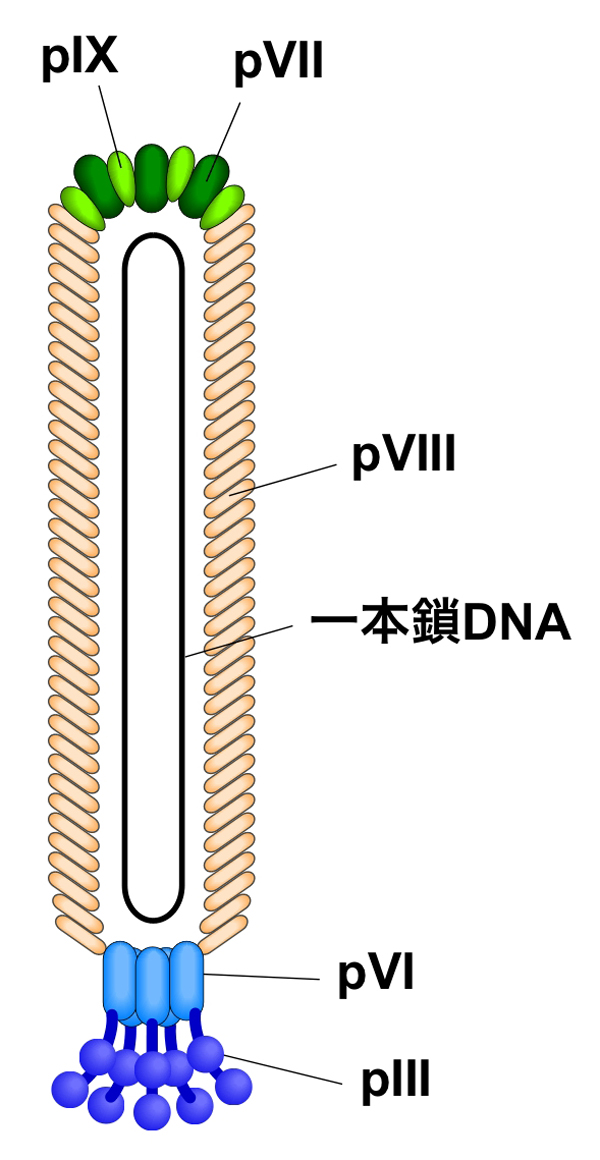
繊維状ファージ
細菌に感染して菌体内で増殖する一群のウイルスをバクテリオファージ(またはファージ)という。バクテリオファージは、その生活環の違いから溶菌性ファージ・溶原性ファージ・繊維状ファージに分類される。
T4ファージやT7ファージなどの溶菌性ファージは、感染後期に宿主を溶菌しながら増殖する。それに対してλファージなどの溶原性ファージは、ファージゲノムが大腸菌の染色体に組み込まれて菌の染色体の一部として増殖するが、誘導因子の添加などによりファージ粒子が形成されると宿主を溶菌して増殖する。
これらに対して繊維状ファージは、溶菌はせずに増殖できる。そして繊維状ファージを用いるメリットは、そのゲノムの大きさにある。T4ファージのゲノムサイズが168,903塩基対、λファージのゲノムサイズが48,502塩基対であるのに対し、繊維状ファージであるM13ファージのゲノムサイズはたったの6,407塩基対(遺伝子はたったの9個)しかない。
繊維状ファージの大腸菌への感染は、大腸菌のF繊毛に結合することで始まり、1本鎖のゲノムDNAが大腸菌内に注入される。感染にはpIIIタンパク質が必須であり、大腸菌のF繊毛とTolA 分子を介して感染する。
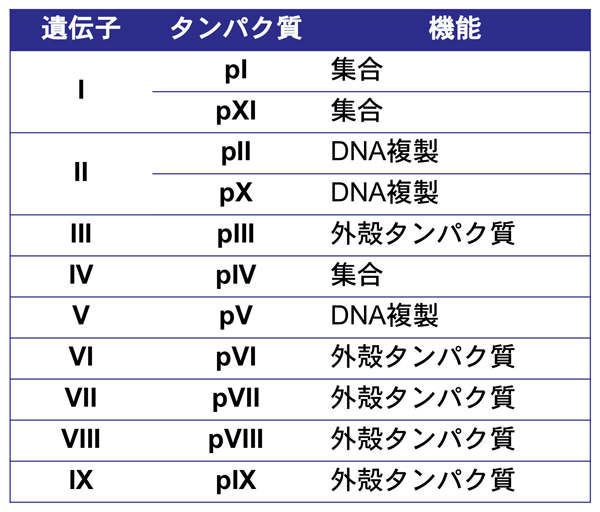
ファージ粒子の形成は大腸菌の外膜および内膜で行われ、粒子の構築と同時に大腸菌の外側へとファージは放出されていく。感染後、新規に複製された一本鎖のゲノムDNAは、pVタンパク質の結合により安定化される。ファージ粒子の構築は、ファージDNA内のパッケージングシグナルにpVIIとpIXが作用することで開始され、外膜のpIV,細胞内膜のpIおよびpXIの多量体はファージ粒子の形成と細胞外に分泌される際の場となり、ファージDNAと内膜に存在するファージの構成タンパク質が結合し細胞外へ放出される。
ファージディスプレイ法
ファージディスプレイ法は、1985年にGeorge P. Smith博士によりが開発された(1)。この最初の報告では、繊維状ファージに制限酵素の57アミノ酸の断片を提示させ、抗体を使った1ラウンドのセレクションにより、この制限酵素の断片を提示するファージを1000倍に濃縮することができた。
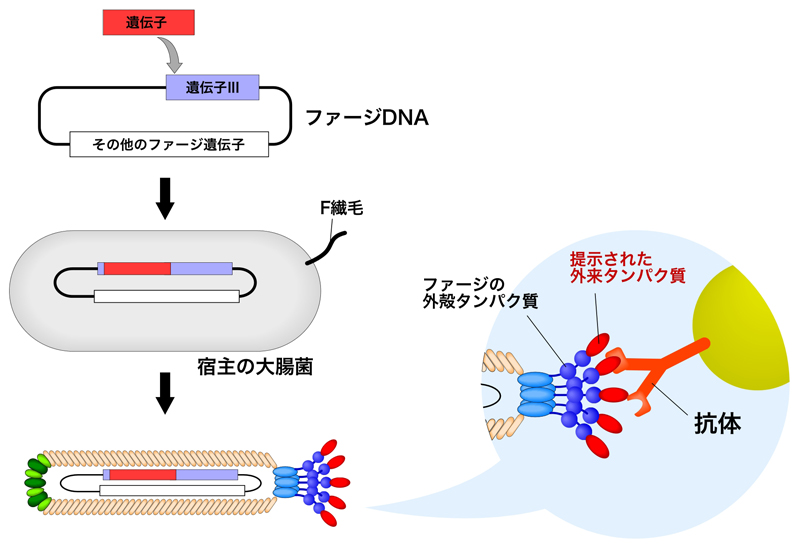
このファージディスプレイに利用されたのは、遺伝子IIIによりコードされるpIIIタンパク質である。pIIIのN末端側には、N1およびN2とよばれる二つのドメインがあり、最初の報告ではN1とN2ドメインの間に制限酵素の断片が挿入された。これにより、繊維状ファジがもつ5つのpIIIすべてが、目的の断片(タンパク質・ペプチド)を提示することになる。こうして、大腸菌への感染能力を維持しつつ、タンパク質を提示させることに成功したのである(でも実は、結構感染に影響していた)。
1988年の論文では、いくつかの改良を加えられた(2)。それは、提示するタンパク質の位置をpIIIのN末端へ移動させた点である。これにより、大腸菌への感染能が大きく改善されたようである。さらに後述するが、バイオパニングという手法が導入された。
ファージディスプレイにはpIIIを使ったものの他、pVIIIを使ったものもある。pVIIIは繊維状ファージの表殻に約2700個も規則正しく並んであるため、これらに提示させることが可能になる。ただし、pVIIIを使った場合、提示できるのは5〜8アミノ酸の短いペプチドに限られる。また、T4ファージ, T7ファージ, λファージを使ったファージディスプレイも報告されている。
バイオパニング
バイオパニングとは、タンパク質(ペプチド)を提示したファージライブラリーの中から、目的のファージを選別する操作である。この方法では、目的のファージが提示するタンパク質と特異的に結合する分子(抗体など)をビオチン化し、これをストレプトアビジンでコートされたビーズ(ディッシュ)に結合させる。ビーズ(ディッシュ)を使って、目的のタンパク質を提示するファージを結合する分子との親和性により精製する。1988年のSmith博士の論文では、βガラクトシダーゼに対する抗体のエピトープを提示するファージが、バイオパニングにより108倍に濃縮された。
ファージミドベクター
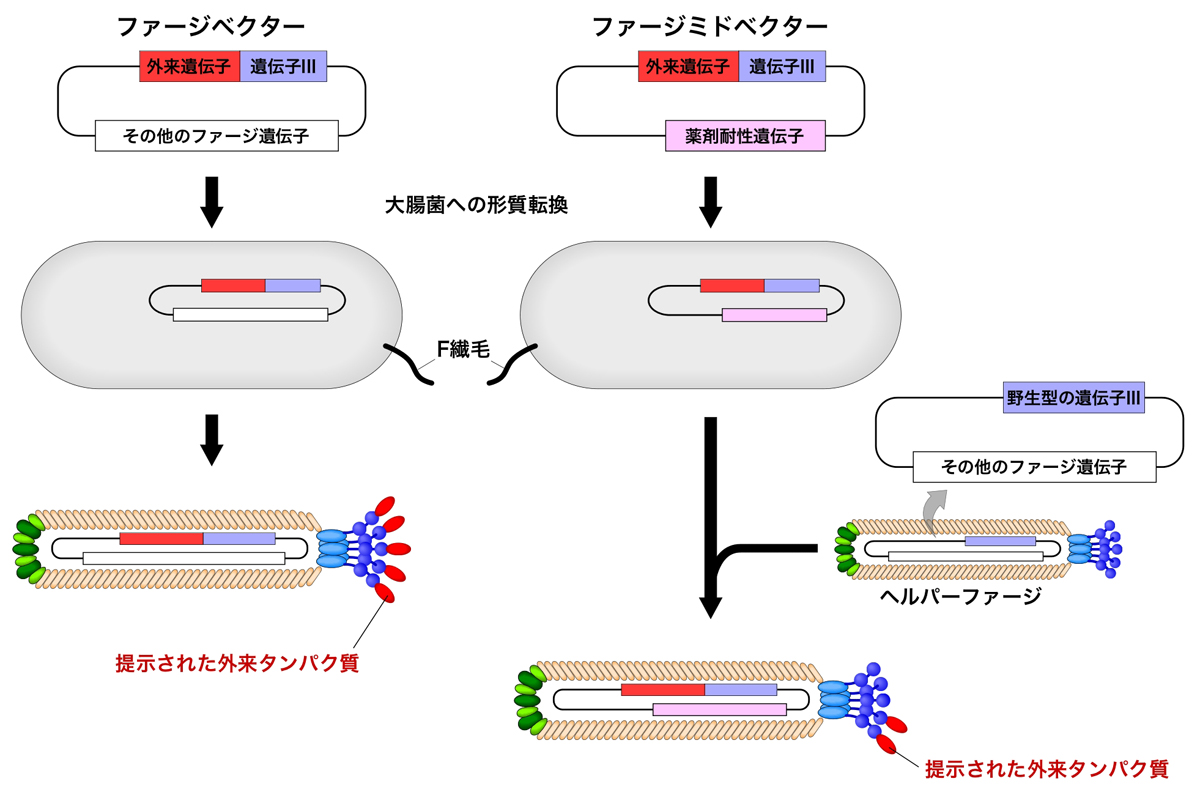
ファージディスプレイには、ファージベクターに加えてファージミドベクターを使った系がある。ファージミドベクターは、プラスミドとしての機能をもつ環状のDNAであり、単独ではファージ粒子を形成できない。
目的のタンパク質をコードする外来遺伝子を遺伝子IIIと融合させた遺伝子をもつファージミドベクターを形質転換させた大腸菌に、ファージ粒子の形成に必要な遺伝子を供給するヘルパーファージ(M13KO7など)を感染させることで、目的のタンパク質を提示させたファージ粒子を作製することができる。この際、目的のタンパク質とpIIIの融合タンパク質は、ヘルパーファージから供給される野生型pIIIと競合しながらファージ粒子に組み込まれるため、目的のタンパク質の提示はpIIIの一部、つまり5つのpIIIのうち1〜3個程度に限られる。しかも、残存する野生型pIIIの存在によって大腸菌への感染能は担保され、さらに大きなタンパク質の提示が可能となる。
また、ヘルパーファージのパッケージングシグナルには変異が導入されており、この方法で作製したファージの中には、ファージミドベクターが優先的にパッケージングされるため、目的のタンパク質の遺伝子はファージ内に格納される。
ファージミドベクターを使うことによって大腸菌への感染能が担保されるが、それ以外にもメリットがある。5つすべてのpIIIにタンパク質を提示するよりは、1〜3個の少数に提示させた方が、選別の際により親和性の高ものを選別しやすくなる。逆に、可能性のあるものを多数選別したいなら、5つのpIIIすべての提示した方がよいだろう。
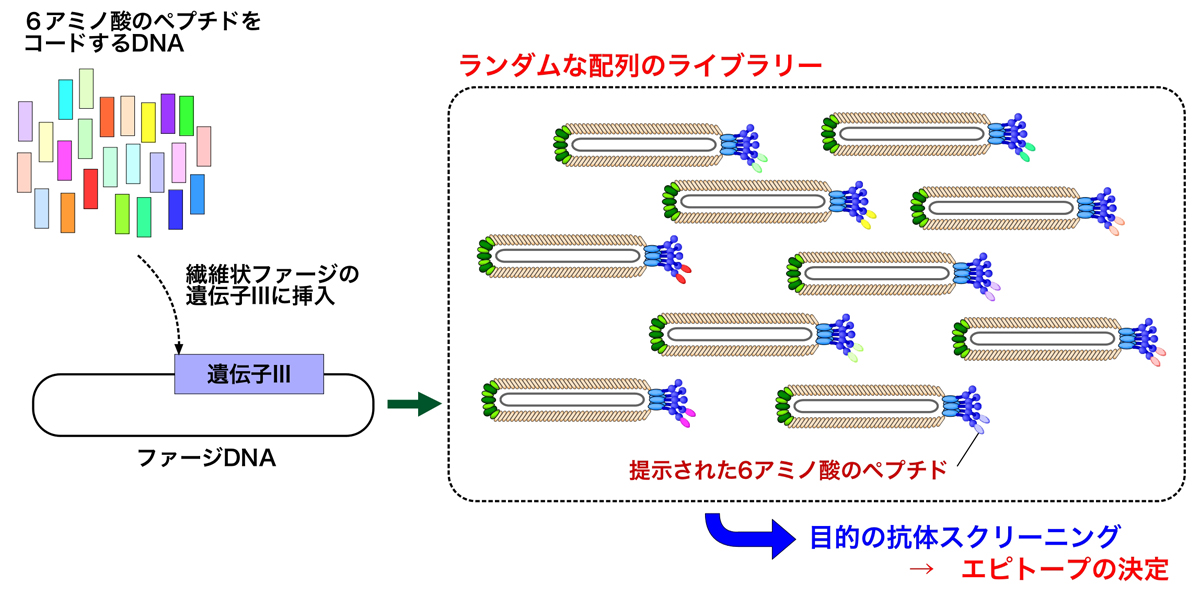
ランダムな配列のディスプレイ
1985年のSmith博士の論文で、ランダムなペプチド断片をもつライブラリーから、抗体によって目的のペプチドを提示するファージを精製できると予想されていた。そして1988年の論文では、ランダムペプチドを提示するファージディスプレイは、抗体によって認識されるエピトープの発見に有用であると書かれていた。
多くの抗体は、5-6アミノ酸残基を認識する。そこで、1990年にSmith博士らは4000万種類の6-merペプチドのライブラリーを作製した(3)。さらに、このライブラリーを使って3ラウンドのバイオパニングと大腸菌への感染を繰り返し、ミオヘムエリトリン(myohemerythrin)の6残基断片を使って作製された2つのモノクローナル抗体のエピトープを決定したのである。
同じく1990年、Devlinらは2000万種の異なる15-merペプチドのライブラリーを報告し、ストレプトアビジンのビオチン結合部位に対する高親和性を与えるペプチド結合モチーフHPQを導き出した(4)。
参考文献
1) Smith, G.P. (1985) Science 228, 1315-1317
2) Parmley, S.F. and Smith, G.P. (1988) Gene 73, 305-318
3) Scott, J.K. and Smith, G.P. (1987) Science 249, 386-390
4) Devlin, J.J., Panganiban, L.C. and Devlin, P.E. (1990) Science 249, 404-406