大腸菌にプラスミドを導入して形質転換体を得たら、その形質転換体を液体培地で培養し、そこからプラスミドを抽出する。これは、遺伝子を扱う研究では必ず行う作業である。市販のキットもいろいろあるが、どれも基本はアルカリ-SDS法を利用したものである。ここでは、アルカリ-SDS法の原理を説明しよう。
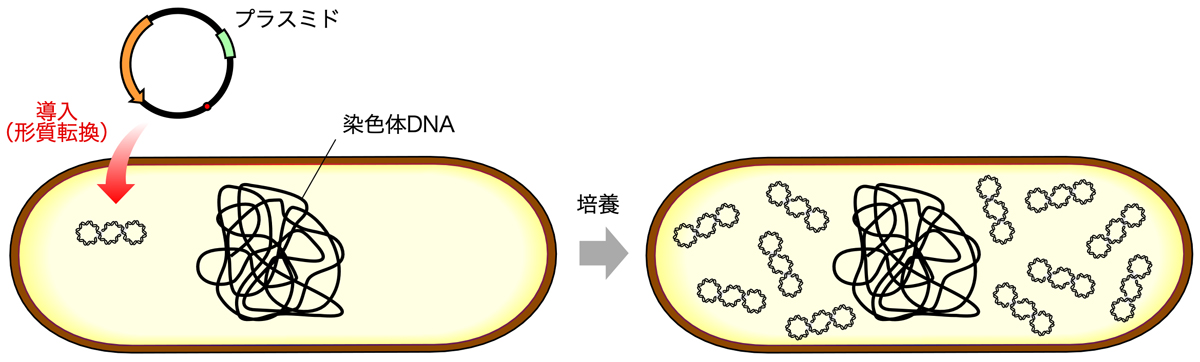
大腸菌とプラスミド
大腸菌に導入されたプラスミドは、細胞内で複製されてそのコピー数を増やしていく。細胞内でのコピー数は、プラスミドがもつ複製起点のタイプによって決まるが、多いものでは1個体あたり約500コピーにもなる。したがって、培養により増殖した大腸菌(形質転換体)からは、大量のプラスミドを抽出することが可能である。
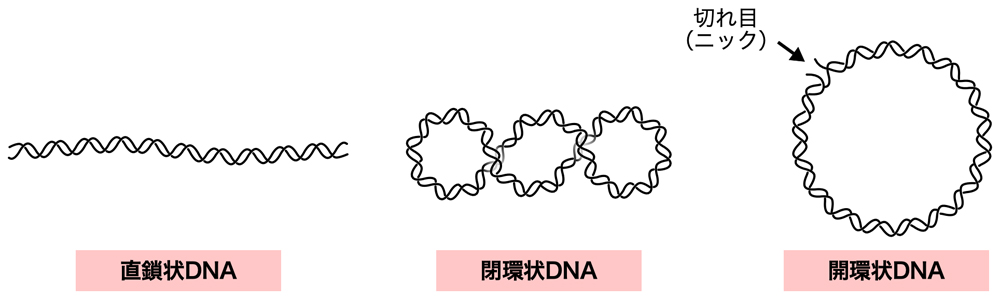
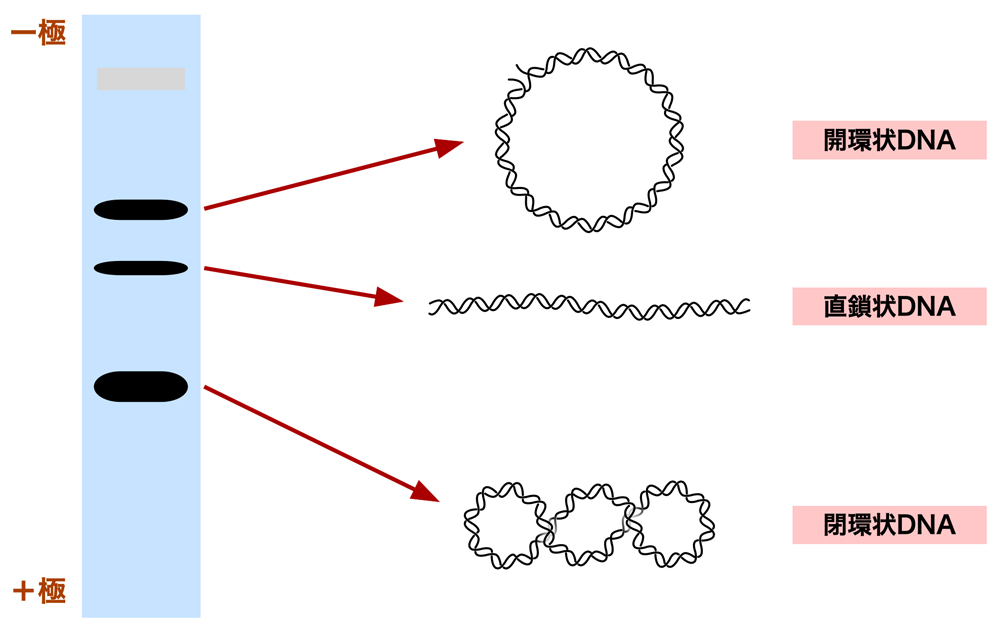
プラスミドは小型の環状DNAであるが、基本的に3つの形状があり得る。大腸菌内にあるプラスミドは閉環状DNAであり、二重らせん構造のDNAが完全に環状に閉じた状態となっている。さらに、細胞内では超らせん(スーパーコイル)とよばれるねじれも入っているため、下図のようなねじれた状態となっている。同じ閉環状DNAでも、超らせんのない弛緩型もある。閉環状DNAの片方のポリヌクレオチド鎖に切れ目(ニック)が入ると、開環状DNAとなる。通常、DNAの二重らせん構造は自由に回転できないが、一方の鎖に切れ目が入ると、もう一方の鎖を軸に切れ目の両側が自由に回転できるようになる。したがって、保持されていた超らせんのねじれはすべて解消され、弛緩型と同じ状態となる。そして両方の鎖に切れ目が入ると、DNA鎖は完全に切断され、直鎖状DNAとなる。
アルカリ-SDS法の手順と原理
はじめに、形質転換によって得られたコロニーを1つ選んで液体培地に移し、振盪培養する。コロニーは、プラスミドを導入された一匹の大腸菌(形質転換体)が増殖して形成されたものなので、1つのコロニー中の大腸菌はすべて同一のプラスミドをもっているはずである。そして、そこから培養によって増殖した大腸菌も、すべて同じプラスミドをもっているはずである。
そこで、大腸菌の培養液をチューブに移して遠心分離し、大腸菌をペレットにする。そして、この大腸菌をグルコース溶液に懸濁する。このグルコース溶液には、pHを中性付近に保つためのTris(緩衝液)、Mg2+を必要とするDNA分解酵素を阻害するためのEDTA(Mg2+やCa2+と結合するキレート剤)が含まれる。
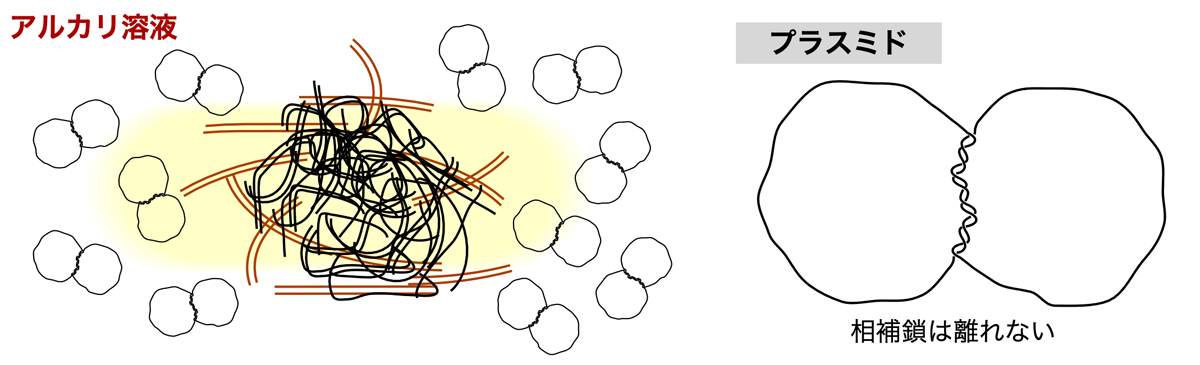
次に、NaOH(水酸化ナトリウム)とSDS(ドデシル硫酸ナトリウム:陰イオン性の界面活性剤)を含むアルカリ溶液を加えて穏やか転倒混和する。激しく撹拌すると染色体DNAが細断されてしまい、その断片がプラスミド溶液に混入することになるので、アルカリ溶液を加えた後は穏やかに撹拌する。大腸菌をアルカリ性の溶液中でSDS処理すると、細胞壁が壊れ、染色体DNAとタンパク質は変性し、プラスミドは溶液中に放出される。アルカリ性の条件では、DNAは変性して2本の鎖が解離する。しかし閉環状DNAは、2本の鎖が互いに巻きついた状態になっているため、変性して塩基対は解離するものの、2本の鎖は互いに離れることない。このとき、大腸菌のタンパク質や壊れた細胞壁や変性した染色体DNAは互いに絡まって巨大な複合体を形成し、それがドデシル硫酸にコートされた状態で溶液中に溶けている。したがって、粘性の高い黄色みがかった透明な溶液となる。
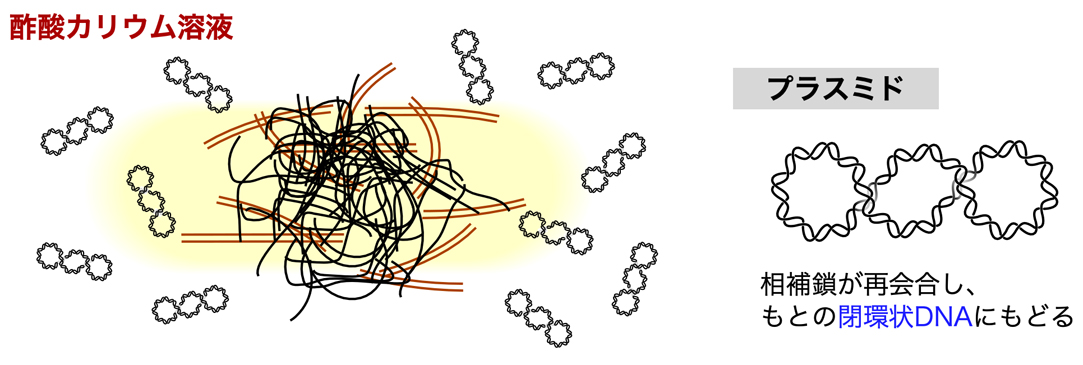
そこで、酢酸カリウム溶液(pH 5.5)を加えてゆっくりと転倒混和する。これによって溶液は中和され、2本の鎖が離れなかったプラスミドは、もとの閉環状DNAに戻ることができる。またここで、タンパク質や細胞壁や染色体DNAが絡まった複合体をコートしていたSDS(ドデシル硫酸ナトリウム)のナトリウムイオン(Na+)が、カリウムイオン(K+)に置換され、この複合体が不溶化する。ECHA (European Chemical Agency)のデータによると、SDSの水への溶解度は20°Cで >130 g/Lである一方、カリウム塩の溶解度は25°Cで415 mg/Lであるらしい。こうして、牛乳に図を混ぜたような状態になる。
これを遠心分離すれば、タンパク質や細胞壁や染色体DNAが絡まった複合体を沈殿として取り除くことができ、上清の溶液中に残ったプラスミドを回収できる。
さらなる精製
あとは、プラスミドを精製するだけである。大腸菌には、プラスミド以外にも染色体DNAやRNA・タンパク質も含まれているが、染色体DNAとタンパク質は上記のアルカリ-SDS法によりある程度除かれる。一方、RNAはプラスミドと同じ溶液中に含まれたままである。市販のプラスミド抽出キットでは、グルコース溶液にRNA分解酵素(RNase)を加えておいてRNAを分解し、アルカリ-SDS法の中和反応後にプラスミドDNAをシリカ膜や陰イオン交換樹脂(DEAE:ジエチルアミノエチル)などを使って精製する。しかしここでは、もっとマニュアルな方法について少し補足しておく。
しっかりタンパク質を除去するのであれば、酢酸カリウム溶液で中和後の上清を用いてフェノール/クロロホルム抽出を行えばよい。簡単に精製するのであれば、酢酸カリウム溶液で中和後の上清からエタノール沈殿により精製する(フェノール/クロロホルム抽出とエタノール沈殿については、詳細はこちら)。ただし、中和後の上清にはRNAも含まれるため、沈殿には大量のRNAも含まれることになる。そこで、RNaseを含むTE緩衝液(TEバッファー)に沈殿を溶解すれば、電気泳動の邪魔にならない程度にRNAは分解されるが、結局RNAが溶液中に残るため分光光度計による定量には使えない。
タンパク質とRNAを除去する効果的な方法は、フェノール/クロロホルム抽出と塩化リチウム(LiCl)沈殿とポリエチレングリコール(PEG)沈殿だろう。LiClは強力なRNAの溶解度を低下させる脱水剤であり、高分子のRNAを沈殿として除去できる(プラスミドは上清に残る)。アルカリ-SDS法の中和後の上清からイソプロパノール沈殿によりプラスミドを沈殿させ、TE緩衝液に溶解する。そこに氷冷した5 M LiCl溶液を等量加えて遠心分離すれば、高分子のRNAは沈殿し、プラスミドは上清に残る。あとは、沈殿を70%エタノールで洗浄し、乾燥後にRNaseを含むTE緩衝液に溶解する。ここでさらに、フェノール/クロロホルム抽出とエタノール沈殿を行い、プラスミドを水に溶解する。最後に、PEG沈殿を行う。ポリエチレングリコール(PEG)は、高塩濃度において高分子のプラスミドを選択的に沈殿させるが、短いRNAは沈殿しない。RNAを構成するリボースの2’位にはOH基があるために、ヌクレオチド1個あたりに含まれるOH基がDNAよりも1つ多い。これによってRNAの方が比較的親水性が高くなっているため、PEG沈殿ではRNAが沈澱せず、プラスミドだけが選択的に沈殿するのである。
ここまで全体の流れをざっくり書いているだけなので、詳細なプロトコールはSambrookの『Molecular Cloning: A Laboratory Manual』などを参考にしてもらいたい。
抽出したプラスミドの電気泳動
大腸菌から抽出したプラスミドを電気泳動すると、だいたい下図のようなバンドが得られる。実は、同じ分子量のプラスミドであっても、その形状によって電気泳動における移動度が異なるのである。最も移動度が大きいのは、閉環状DNAである。超らせんによってコンパクトにねじれた構造となっているため、移動度が大きいようである。次に移動度が大きいのが直鎖状DNAで、最も移動度が小さいのが開環状DNAである。弛緩型の閉環状DNAは、開環状DNAと同じ移動度になる。大腸菌内にあるのはすべて閉環状DNAだが、抽出の過程で多少傷ついたプラスミドも生じてしまうようである。また、これら3つのバンドの上にさらに二量体のプラスミドが混入した染色体DNAのバンドが見えることもある。