リボソームを構成するrRNAのうち、5S rRNAはRNAポリメラーゼIIIによって転写される。一方、28S, 18S, 5.8S rRNAの3つはRNAポリメラーゼIによって45S rRNA前駆体として転写され、修飾・切断を受けて成熟したrRNAとなる。この頁では、rRNAの修飾部位を決めるsnoRNAについて紹介しよう。
rRNAの修飾をガイドするsnoRNA
rRNAは、2’-O-メチル化とウリジンの異性化の2種類の化学修飾を受ける。この修飾部位を決めているのが、核小体低分子RNA(snoRNA:small nucleolar RNA)である。snoRNAは、核小体に局在する60~300ヌクレオチドの短いRNAである。
snoRNAには、Box C/DとBox H/ACAの2つの主要なタイプがある。2’-O-メチル化部位を決めるのがBox C/DタイプのsnoRNAであり、ウリジンの異性化部位を決めるのがBox H/ACAタイプのsnoRNAである。また、ここでは扱わないが、リボソームの生合成に必須のRMRPや、テロメアの維持に関わるTERCといったsnoRNAも存在する。
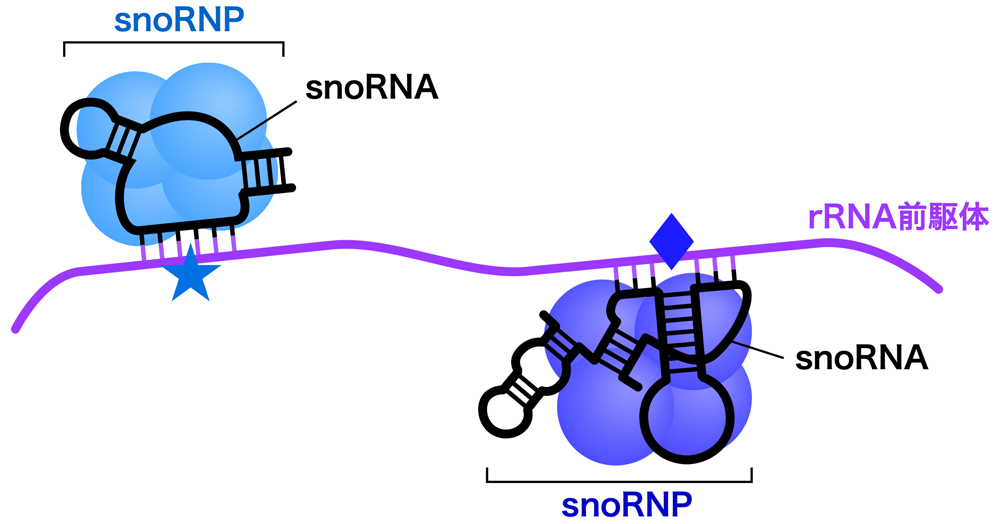
それぞれのsnoRNAはタンパク質と結合し、核小体低分子リボ核タンパク質(snoRNP:small nucleolar RiboNucleoProtein)として機能する。snoRNAが標的RNAと塩基対を介して配列特異的に結合し、修飾部位へガイドする。そして、タンパク質成分が修飾反応を触媒する。
Box C/D snoRNA
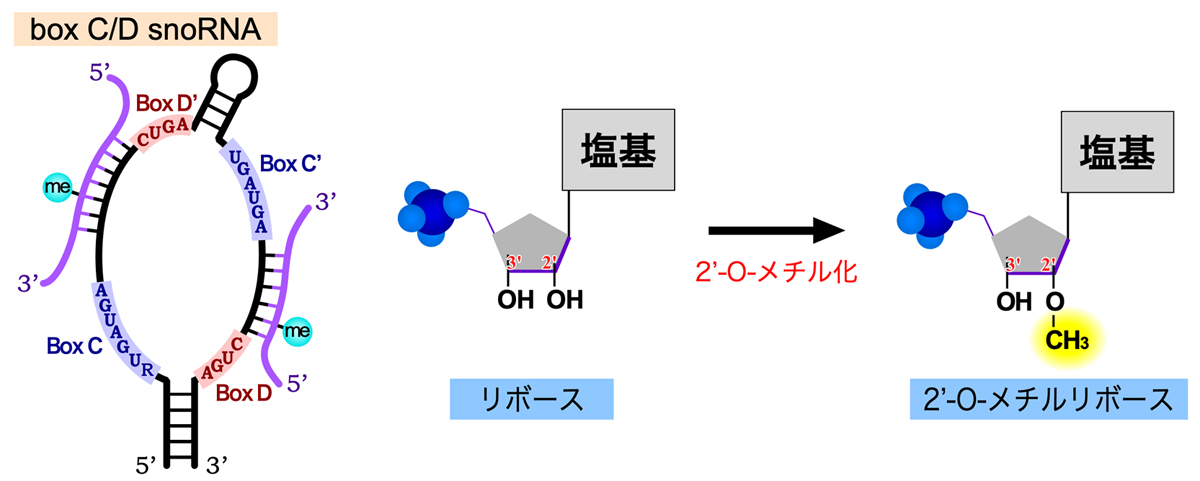
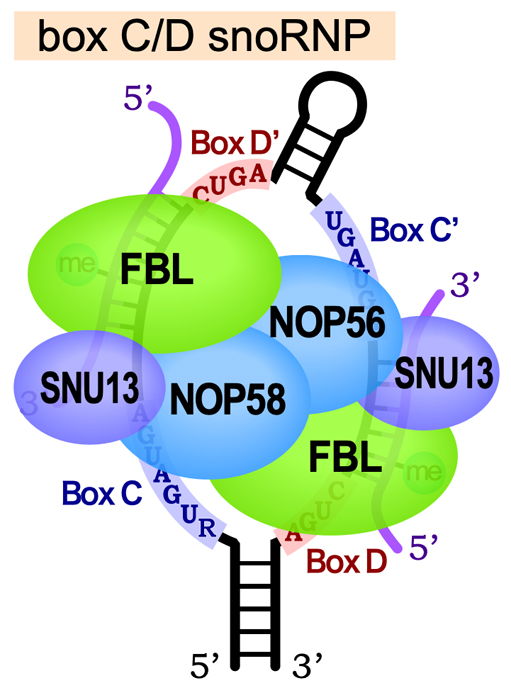
45S rRNA前駆体が受ける化学修飾の1つは2’-O-メチル化である。これは塩基ではなく糖(リボース)の修飾であり、2’位の-OH基をメチル化修飾して-O-CH3とし、2’-O-メチルリボースを形成する。この修飾によりRNAの親水性が低下し、rRNAがリボソーム内に埋め込まれるのに役立つと考えられる。この修飾をガイドするのが、Box C/D snoRNAである。Box C/D snoRNAには保存された配列モチーフBox C(RUGAUGA)とBox D(CUGA)があり、さらに少し保存性が低いもう1セットのBox C’とBox D’もある。そして、これらのBoxを含む内部ループをもったヘアピン構造を形成する。
Box C/D snoRNAは、Fibrillarin (FBL)・SNU13・NOP58・NOP56の4種類のタンパク質と結合してBox C/D snoRNPとなる。この中で、FBLがメチルトランスフェラーゼとしてはたらき、2’-O-メチル化を触媒する。メチル化の際は、Box DとBox D’の5’側で標的RNAと塩基対を形成し、Box D/D’の5 ‘末端から5ヌクレオチドの位置にあるヌクレオチドがFBLにより2’-O-メチル化される。
Box H/ACA snoRNA
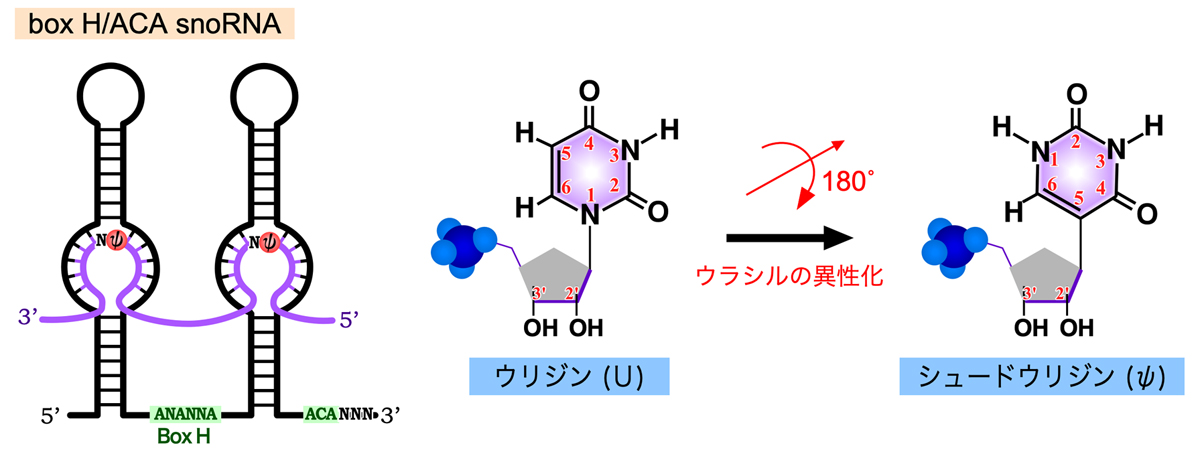
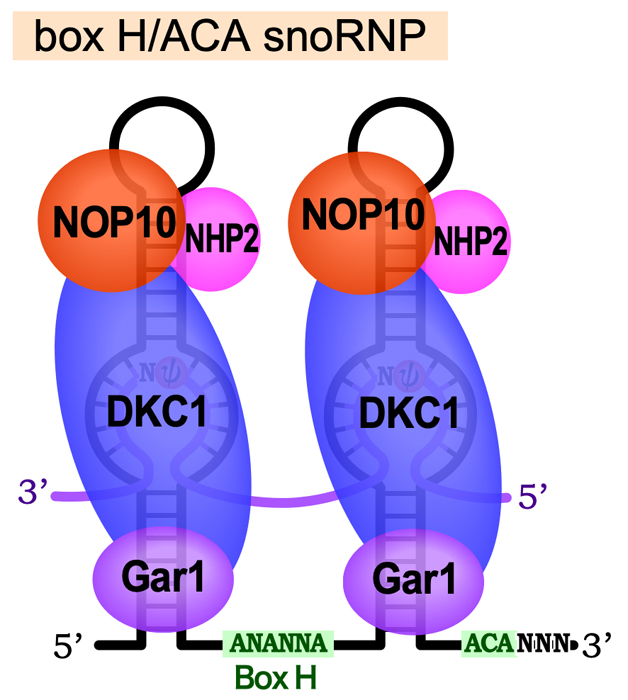
45S rRNA前駆体が受けるもうひとつの化学修飾が、ウリジンの異性化によるシュードウリジン(プソイドウリジンともいう)の形成である。これは、ウラシルを3位と6位を結ぶ軸を中心に180°フリップさせ、5位の炭素(C)でリボースに結合させたものである。この修飾によりウラシルの1位のNが解放されるため、より多くの水素結合を作れるようになり、リボソームの構造の形成・維持に利用されると考えられる。Box H/ACA snoRNAには、Box H(ANANNA)で連結された2つのステムループ構造をもち、3’末端には保存されたACA配列がある。そして、それぞれのステムループには、内部ループが存在する。
Box H/ACA snoRNAは、NHP2・NOP10・Gar1・Dyskerin (DKC1)の4種類のタンパク質と結合してBox H/ACA snoRNPとなる。この中で、DKC1がウリジンイソメラーゼとしてはたらき、ウリジンの異性化を触媒する。内部ループの9〜16ヌクレオチドの領域で標的RNAと塩基対を形成し、Box HおよびACA配列から約15ヌクレオチドの位置にある「シュードウリジン化ポケット」とよばれる部位のウリジン残基が、シュードウリジン化される。
snoRNA遺伝子
snoRNAも遺伝子にコードされている。大腸菌にはsnoRNA(遺伝子)はないが、真核生物においてsnoRNAが獲得され、その後進化(生物の複雑さ)とともに増加している。出芽酵母(Saccharomyces cerevisiae)では64個の転写単位に76個のsnoRNAがコードされているが、ヒトでは2,000個以上のsnoRNAがあると推定されている。
snoRNA遺伝子には、プロモーターから独立に転写されるsnoRNA遺伝子や、プロモーターをもたず他の遺伝子のイントロン内にコードされるsnoRNA遺伝子がある。またどちらの場合にも、モノシストロニック(1つの転写単位に1つのsnoRNAをコード)に発現するものと、ポリシストロニック(1つの転写単位に複数のsnoRNAをコード)に発現するものがある。独立に転写される場合は、大部分のsnoRNA遺伝子がRNAポリメラーゼIIによって転写されるが、RNAポリメラーゼIIIによって転写されるものもある。また、タンパク質をコードするmRNAのイントロン中にもあるし、非コードRNAのイントロン中にもある。
酵母では、多くが独立の遺伝子としてプロモーターから転写されており、モノシストロニックに発現する(ポリシストロニックに発現するものや、イントロンにコードされるsnoRNAも少数ある)。植物でも、多くのsnoRNAが独立の遺伝子としてプロモーターから転写されており、しかもポリシストロニックに発現するものが多い。
動物では、snoRNAはほとんどがイントロン内にコードされており、多くが1つのイントロンあたり1つのsnoRNAとなっている。面白いことに、イントロン内にコードされた多くのsnoRNAが、リボソーム生合成または翻訳装置をコードする遺伝子内に見られるらしく、リボソームタンパク質遺伝子の3分の1以上が、少なくとも1つのsnoRNAをコードしている。また、動物にも独立の遺伝子として存在するU3, U8, RMRPのようなsnoRNA遺伝子もある。
snoRNAの成熟
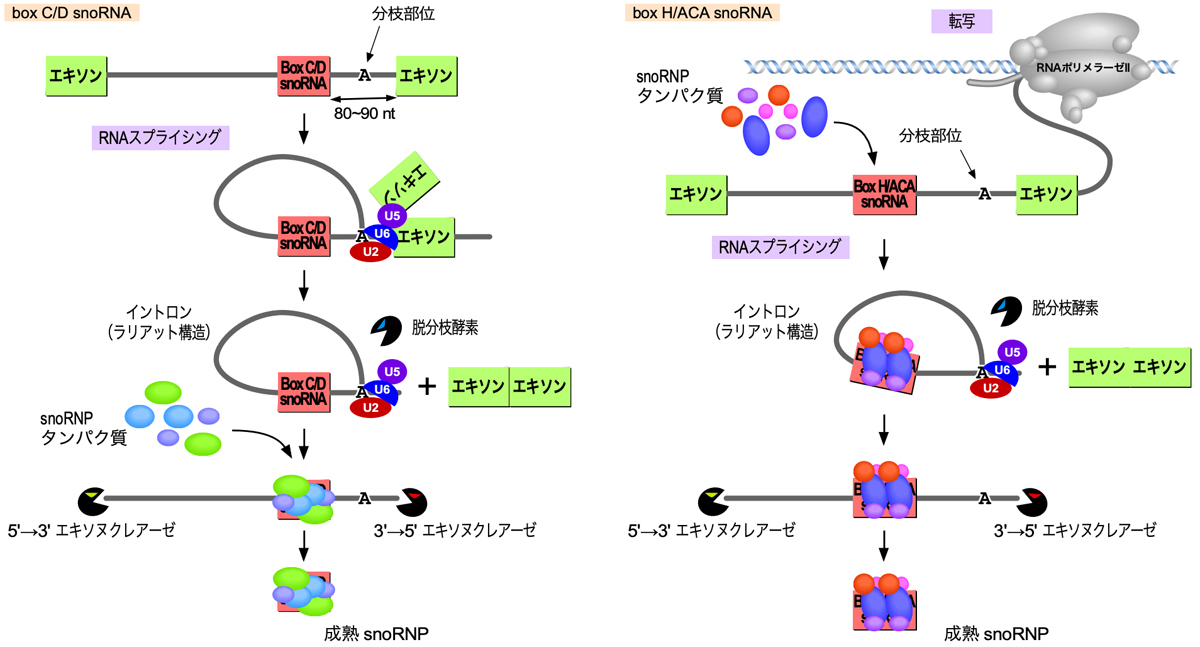
上述の通り、動物では多くのsnoRNAがmRNA前駆体のイントロン内にコードされている。したがって通常、成熟したsnoRNAはRNAスプライシングによって切り出されたイントロンの加工により生成される。そして、snoRNAの末端の形成には、snoRNPタンパク質の結合が重要となる。
ヒトのほとんどのBox C/D snoRNA遺伝子は、イントロン内の3’スプライス部位から上流80〜90ヌクレオチドの位置にある(分枝部位から約50ヌクレオチドの距離がいいらしい)。RNAスプライシングが起こり、C1複合体になるとSNU13がsnoRNAにリクルートされ、その後でFBL・NOP58・NOP56が結合するようである。スプライシングによりラリアット構造としてイントロンが切り出されたのち、脱分枝酵素により直鎖化され、さらに両側からエキソヌクレアーゼによって削られていく。そして、結合したタンパク質がエキソヌクレアーゼによる削り込みから保護することで、snoRNAの末端が決まる。
Box H/ACA snoRNA遺伝子については、イントロン内の位置はランダムなようである。そして、NHP2・NOP10・Gar1・DKC1の4つのsnoRNPタンパク質はmRNA前駆体の転写中もしくは直後に結合するようである。あとは、Box C/D snoRNAと同様にスプライシングに依存してsnoRNAの生成が行われるようである。
独立の遺伝子としてプロモーターからの転写により生成されるsnoRNAも、プロセシングは必要である。また、rRNA前駆体の修飾に限らずさまざまな役割を果たすsnoRNAがあり、ここではずべてをカバーできていなので、これ以上のことは原著論文や総説などを読んで欲しい。