転写の段階には開始・伸長・終結の3段階があるが、今回は終結のお話である。細菌の場合、転写はターミネーターで終わる。また、真核生物にはRNAポリメラーゼが3種類あり、それぞれで転写終結のしくみが異なる。しかも、RNAポリメラーゼIIによる転写には明確なターミネーターがない。そこで、細菌と真核生物の転写終結のしくみの違いについて説明しよう。
細菌のρ因子非依存性転写終結
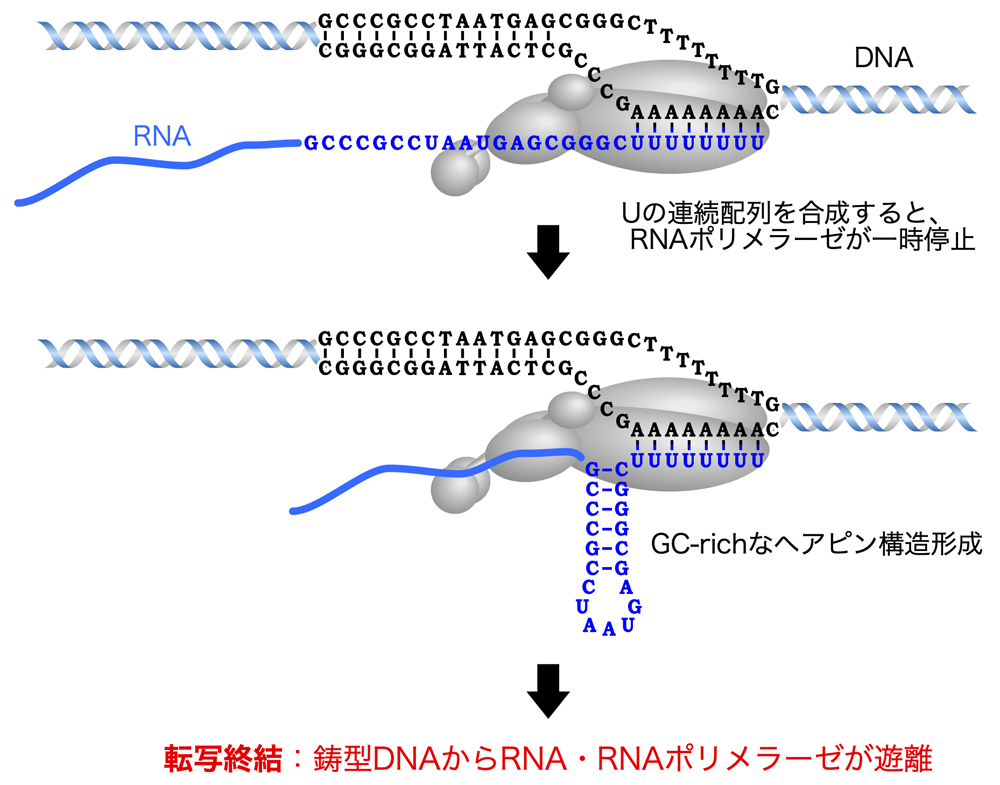
『転写の基礎』の頁でも説明したが、細菌の遺伝子の転写はターミネーターで終わる。細菌のターミネーターは、転写終結にρ(Rho)因子を必要とするかどうかで、2つに分類される。ρ因子を必要としないのが、ρ因子非依存性ターミネーター(rho-independent terminator)であり、内因性ターミネーター(intrinsic terminator)ともよばれる。ここでは、内因性ターミネーターの名前で説明していく。
内因性ターミネーターはDNA中に存在している配列であり、GC-richな逆方向反復配列とそれに続く7〜8個のTの連続配列(相補鎖ではAの連続配列)で構成される。Tの連続配列はよく保存されており、中でも最初の3つのTが最もよく保存されている。一方、GC-richな逆方向反復配列については、GC-richな逆方向反復配列であること以外は保存されてない。RNAポリメラーゼが遺伝子を転写し、mRNAの3’末端付近のターミネーター領域を転写すると、GC-richな逆方向反復配列とそれに続くUの連続配列が合成される。この領域のRNAが下図のようなヘアピン構造を形成することで、ターミネーターとして機能する。
RNAポリメラーゼがUの連続配列の最後のヌクレオチドを合成すると、DNA:RNAハイブリッドの不安定な塩基対によりRNAポリメラーゼが一時停止する。RNAポリメラーゼが停止すると、RNAの出口のところでGC-richな逆方向反復配列によりヘアピン構造が形成される。あとは、RNA鎖の伸長なしでポリメラーゼを前進させるか、あるいはRNA鎖を引っ張ることでUの連続配列でのDNA:RNAハイブリッド領域を短く不安定にし、RNAポリメラーゼを解離させるようである。
ρ因子依存性転写終結
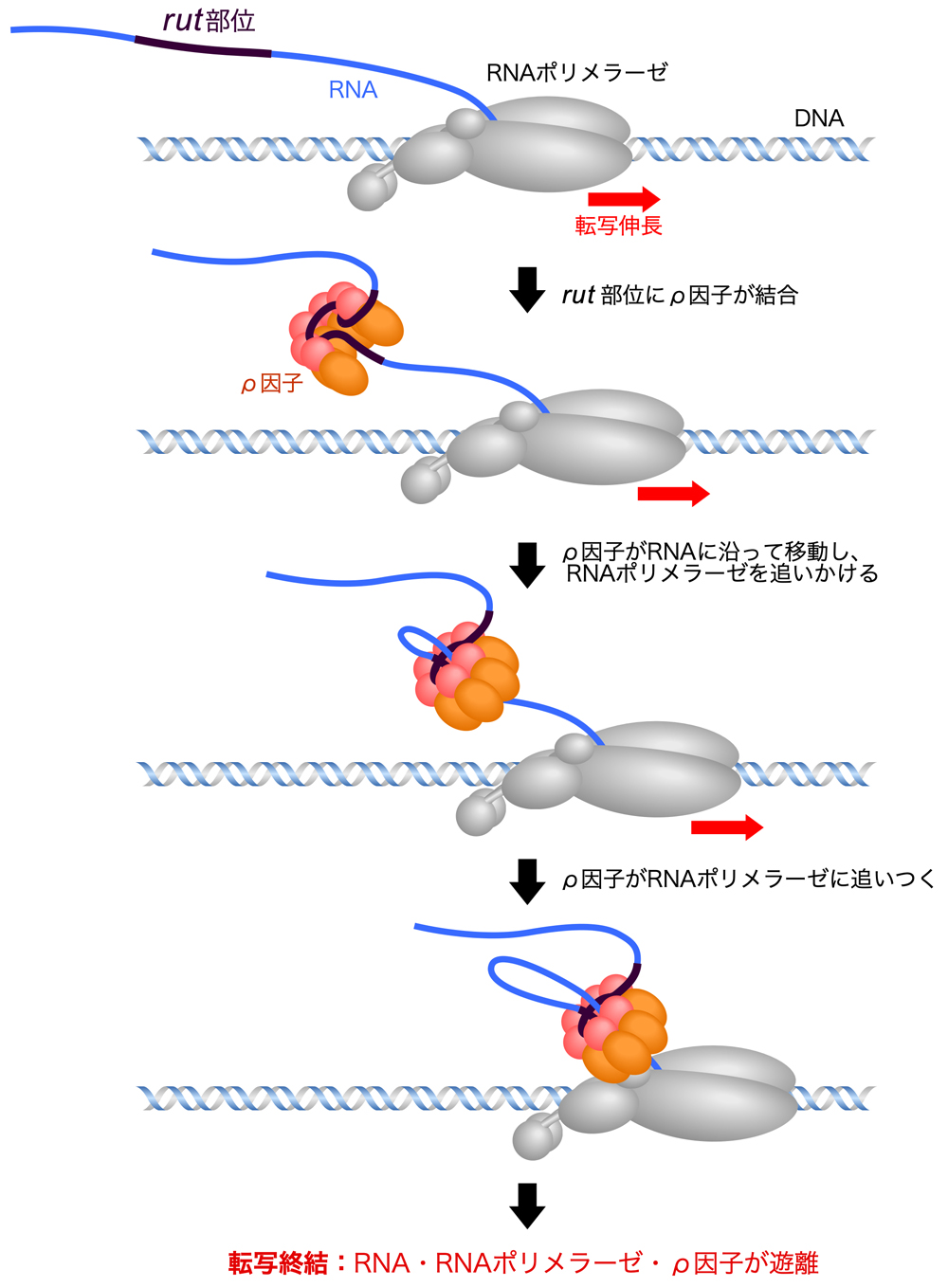
細菌におけるもうひとつのターミネーターが、終結にρ因子を必要とするρ因子依存性ターミネーター(rho-dependent terminator)である。ρ因子とは、ATPの加水分解によって得られるエネルギーを必要とするヘリカーゼであり、ホモ6量体で転写後のRNAに対して作用する。まず、ρ因子はターミネーターよりも上流のRNAに結合し、そこからRNAに沿って移動し、RNAポリメラーゼを追いかける。RNAポリメラーゼはターミネーターで立ち止まり、そこにρ因子が追いつくと、転写が終結する。ρ因子は、RNAポリメラーゼのところで形成されているDNA-RNAハイブリッドの部分に入り込み、これによって鋳型DNAからRNAポリメラーゼやRNAが遊離し、転写が終結する。
ρ因子が結合するのは、RNA上のターミネーターの上流にあるrut (Rho Utilization) 部位である。rut部位は、シトシン(C)が圧倒的に多くてグアニン(G)が少なく、特定の二次構造をとりにくいような約80〜90ヌクレオチドの配列である。通常、終結部位はrut部位からさらに10〜20ヌクレオチド以内にあり、100ヌクレオチドを超えることは滅多にない。
RNAポリメラーゼによりrut部位が転写されると、rut部位にρ因子の6つのサブユニットが結合する。このとき、RNA鎖が通る通路を確保するために、開いたリング構造となる。その後リングが閉じて活性型の酵素となり、ATPのエネルギーを利用してRNAに沿って移動し、RNAポリメラーゼを追いかける。ρ因子はRNAポリメラーゼの2〜5倍速く移動するため、すぐにRNAポリメラーゼに追いつく。その後、ρ因子がその強力なモーターによりRNAを転写複合体から引き抜き、転写は終結する。
真核生物の転写終結
真核生物には3種類のRNAポリメラーゼが存在する。RNAポリメラーゼIIによる転写の場合、明確なターミネーターとよばれる配列はない。しかも、ターミネーターによる転写終結部位がmRNAの3’末端となる細菌と異なり、真核生物ではmRNAの3’末端と転写終結部位は一致しない。
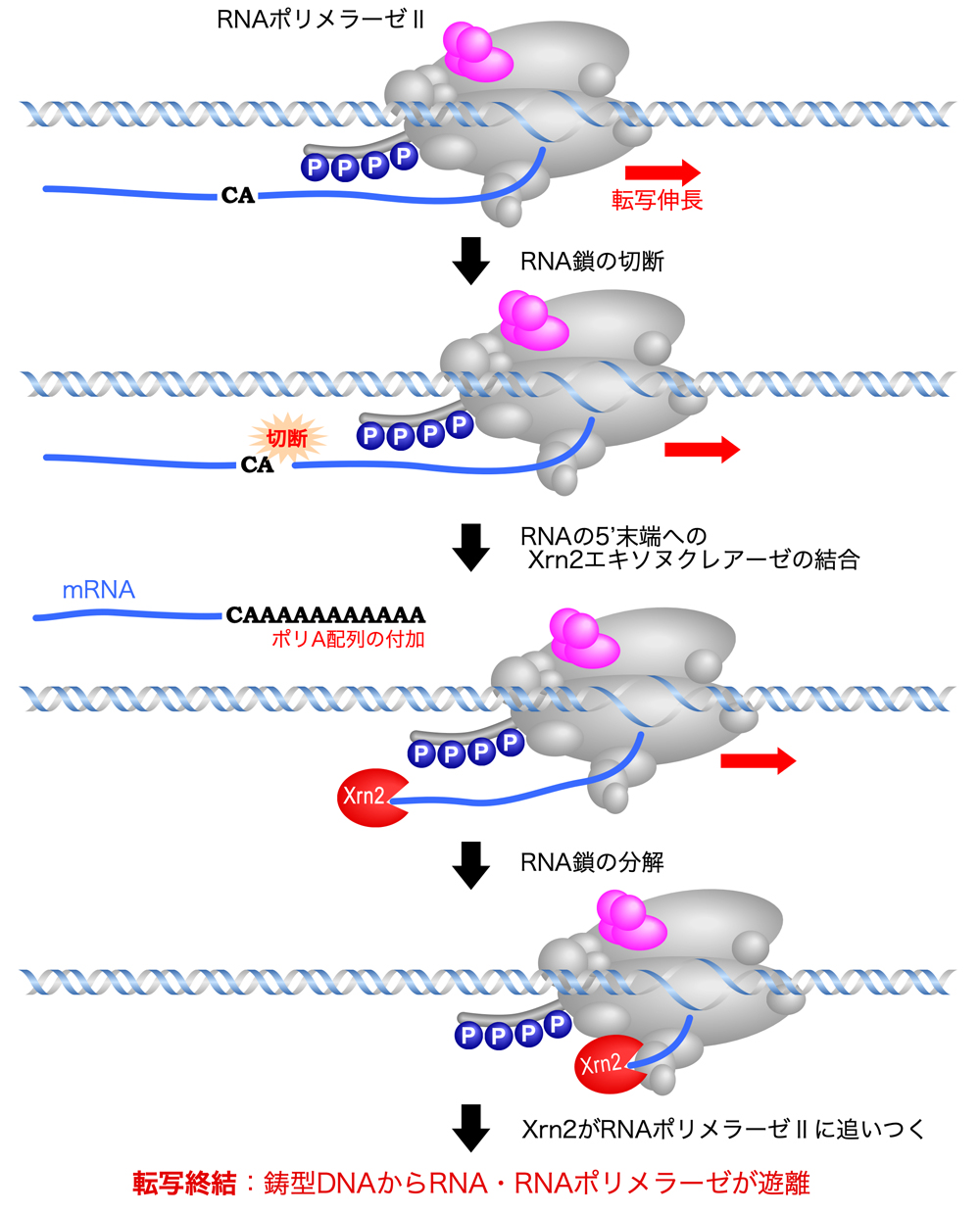
RNAポリメラーゼIIにより3’末端形成のシグナル(ポリAシグナルとGU-rich配列)が転写されると、CA配列の3’側でRNA鎖が切断され、切断により生じた3’末端にポリA配列が付加される(詳細は、RNAプロセシングの頁で)。こうしてmRNAが完成して放出されるが、RNAポリメラーゼはさらに転写を継続している。
切断によって生じたキャップ構造のないRNAの5’末端に5’→3’エキソヌクレアーゼ(酵母ではRat1、ヒトではXrn2)が結合すると、エキソヌクレアーゼはこのRNAを5’末端から分解していく。そして、エキソヌクレアーゼがRNAポリメラーゼIIに追いついたところで、転写は終結する。
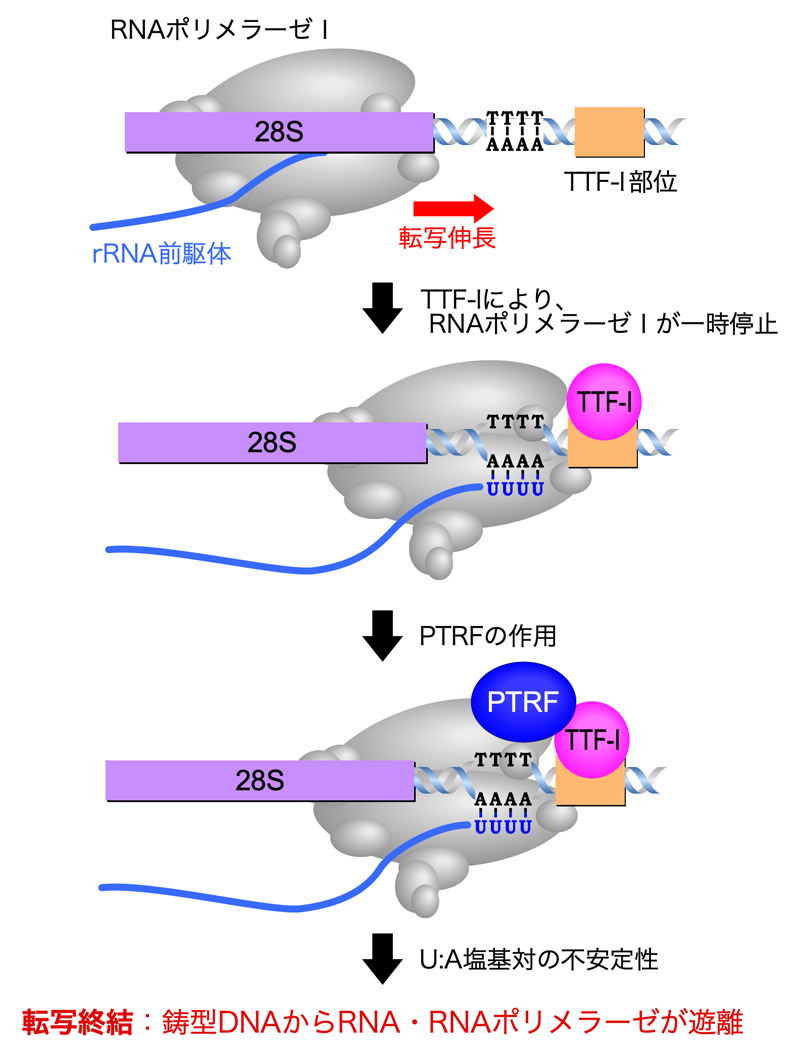
RNAポリメラーゼIによる転写の場合、転写終結はT-rich配列とそれに続くMybファミリーのDNA結合タンパク質の認識配列により起こる。まず、転写しているRNAポリメラーゼIはDNAに結合したMyb様因子によって邪魔され、一時停止する。哺乳類では、RNAポリメラーゼをI一時停止させるMyb様タンパク質はTTF-I(Transcription termination factor 1)である。その後、RNAポリメラーゼIに結合しTTF-Iと直接相互作用するPTRF(polymerase I and transcript release factor)の作用によってRNAポリメラーゼの放出が促進される。鋳型DNAからのRNAポリメラーゼIの放出にはT-rich配列の存在が必要で、合成されたU-rich配列でのRNA:DNAハイブリッド(U:A塩基対)の不安定性により、一時停止した伸長複合体も不安定化することが重要らしい。
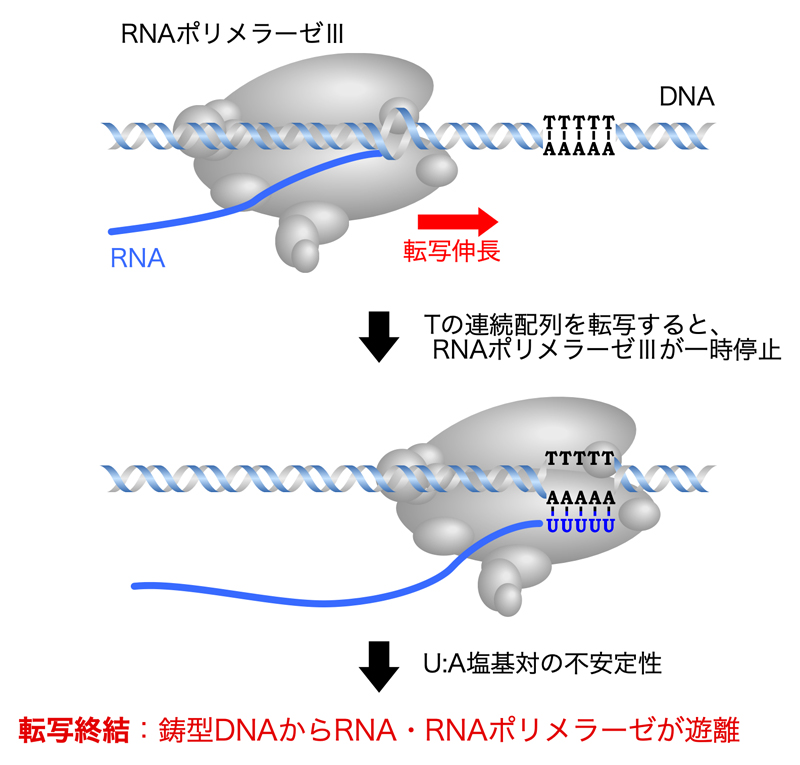
真核生物のRNAポリメラーゼIIIによる転写の場合、他の因子を必要とせずに、鋳型DNAにおけるTの連続配列で効率的に転写を終了できる。酵母では、ほとんどのRNAポリメラーゼIIIのターミネーターに6個または7個のTが含まれるが、哺乳類では4個または5個のTより長いターミネーターはあまりない。RNAポリメラーゼIIIはTの連続配列を転写すると一時停止し、その後RNA:DNAハイブリッド(U:A塩基対)の不安定性によりRNAポリメラーゼIIIによる転写が終結する。RNAポリメラーゼIIIは、RNA:DNAハイブリッド(U:A塩基対)に対する感受性がとくに高いようである。また、細菌の内因性ターミネーターのように、RNAポリメラーゼIIIがTの連続配列で効率的に一時停止し、上流でのヘアピン構造の形成に依存して終結する場合もあるようである。