ゲノム編集について、その応用技術をいくつか紹介しょう。
ゲノム編集のツールには、ZFN, TALEN, CRISPR-Cas9が使われるが、どれにもその派生技術・応用技術がある。ただ、特定の塩基配列を認識させるツールを作製するのに、結合モジュールを連結させて組み立てる必要があるZFNやTALENに比べて、同一のCas9タンパク質に対してガイドRNAだけを変えれば標的配列も変えられる簡便さから、CRISPRの方が応用の幅も広くなる。そこでこの頁では、CRISPRを中心にその応用技術を紹介していこう。
改変型のCas9
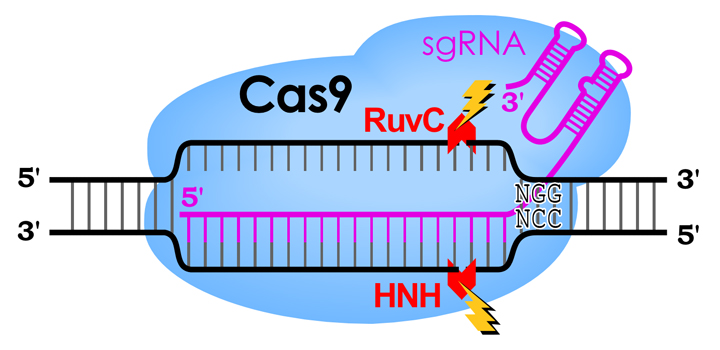
CRISPR-Cas9システムでは、ガイドRNA(sgRNA)と相補的な配列をもつDNAにCas9タンパク質が結合し、その結合部位が切断される。Cas9には、2つのヌクレアーゼドメインがある。1つはsgRNAが直接結合する標的鎖を切断するHNHドメイン、もう1つは非標的鎖を切断するRuvCドメインである。これら2つのヌクレアーゼドメインでDNAのそれぞれの鎖に切れ目を入れることにより、DNA二本鎖切断(DSB)を生じる。
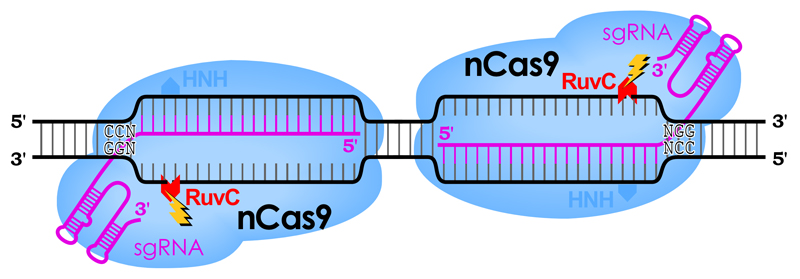
これらのドメインのうち、どちらか一方に変異を導入してヌクレアーゼ活性を消失させると、ニッカーゼCas9(nCas9; nickase Cas9)となり、DNAの一方の鎖にのみ切れ目(ニック)を入れることができる。このnCas9をペアで使うと、それぞれのnCas9が近接するそれぞれのDNA鎖に切れ目を入れ、結果としてDSBを導入することになる。2つのnCas9が揃ってDSBを誘導することで、より特異性の高い編集が可能となる。
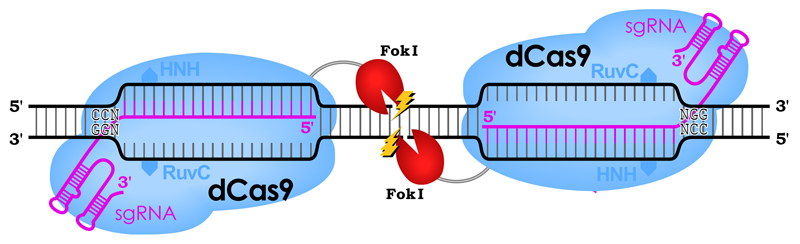
Cas9の両方のヌクレアーゼドメインに変異を導入すると、不活性型Cas9(dCas9; dead Cas9 or deactivated Cas9)となる。このdCas9自体にはヌクレアーゼ活性はないが、ZFNやTALENと同様にFokIヌクレアーゼと連結してdCas9-FokIとし、これをペアで使えばnCas9のように特異性の高い編集が可能となる。
さらに、dCas9を別の因子(転写因子・修飾因子・蛍光タンパク質)と連結すれば、ゲノム機能を調節できる様々なツールとして利用可能となる。
dCas9で遺伝子を調節する
dCas9を使えば、人工転写調節因子を作製して遺伝子の転写の調節も可能となる。最もシンプルな使い方は、dCas9を立体障害として使い単純に転写装置(RNAポリメラーゼ)の邪魔をすればよい。このような使い方をCRISPR干渉(CRISPRi; CRISPR interference)という。CRISPRiは、非鋳型鎖(センス鎖)を標的とした方が効果が高いようである。さらに、CRISPRiはバクテリアでは効率よく転写抑制できるが、哺乳類細胞ではdCas9と転写抑制ドメイン(例えばKox1のKRABドメイン)を融合させるとさらに効果的に抑制できることが報告されている。
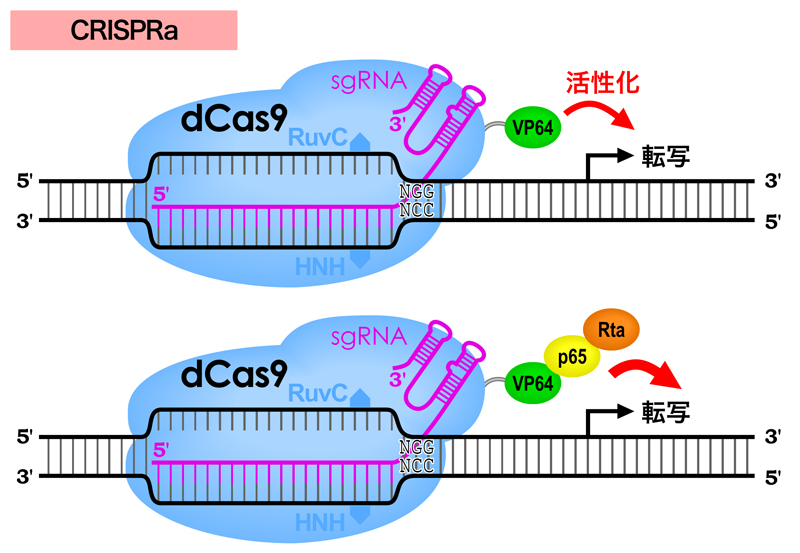
逆に、dCas9を転写活性化因子(例えばVP64やp65)と融合して、人工転写活性化因子として使うこともできる。このような使い方をCRISPR活性化(CRISPRa; CRISPR activation)という。また、VP64とp65とRtaの3つ(VPR)をdCas9にタンデムに連結することにより、さらに強力に活性化することも可能となる。
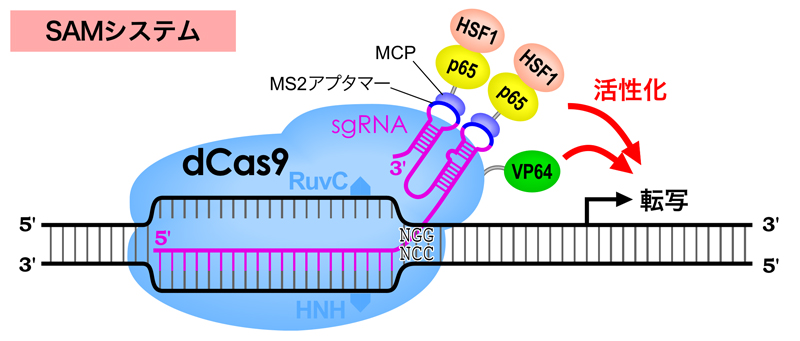
さらに、転写活性化因子あるいは抑制因子をdCas9と直接融合するのではなく、これらが結合する土台をdCas9に用意することができる。例えば、SAMシステム(synergistic activation mediator)では、dCas9にはVP64を融合しておき、それとは別にバクテリオファージMS2のカプシドタンパク質(MCP)と結合するMS2アプタマーをsgRNAのループ部分に付けておく。そして、MCPとp65とHSF1活性化ドメインを融合した因子を共発現させると、MCP-p65-HSF1融合因子がsgRNAのMS2アプタマーにリクルートされ、dCas9-VP64が結合した領域(遺伝子)を効果的に活性化できるのである。
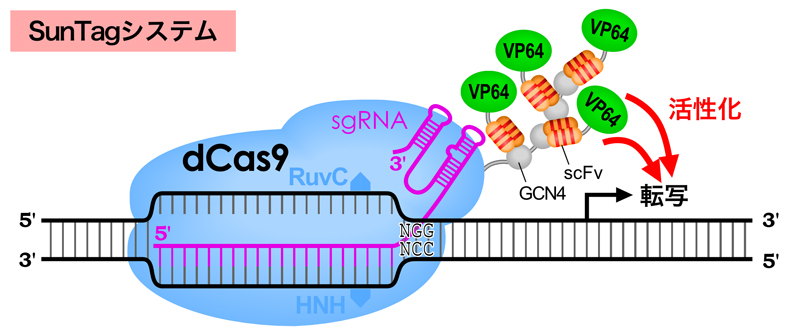
同様に、SunTagシステムでは、複数のGCN4エピトープ(SunTag)をdCas9と融合させておく。また、GCN4エピトープと特異的に結合するscFv(抗GCN4抗体の可変領域VHとVLをリンカーで直鎖状に連結させたもの)とVP64の融合因子を作製し、これをdCas9-SunTagと共発現させると、scFv-VP64融合因子がdCas9に連結したSunTagにリクルートされ、効果的に転写を活性化できるのである。
dCas9でエピゲノム編集
ゲノムは、DNAの塩基配列の情報である。遺伝子の発現(転写)は、特定の塩基配列に転写活性化因子や転写抑制因子が結合することによって制御される。一方、転写はDNAやヒストンの化学修飾によっても制御を受ける。このようなゲノムに加えられる化学修飾の情報を、エピゲノムとよぶ。つまり、ゲノムの塩基配列を編集するゲノム編集に対して、エピゲノム編集はゲノムの修飾状態(エピゲノム)を編集するのである。
エピゲノムを改変するといえば、抗がん剤としても使われるアザシチジンがある。アザシチジンはDNAのメチル化を阻害する効果をもつが、ゲノム全体に作用し、標的とする遺伝子を制限することはできない。一方エピゲノム編集であれば、標的とする遺伝子のエピゲノム状態のみを編集することができる。
実際に、DNAメチル基転移酵素Dnmt3A、DNA脱メチル化酵素TET1、ヒストン脱メチル化酵素LSD1、ヒストンアセチル基転移酵素p300の触媒ドメインなどが修飾因子として使われている。上記のSAMシステムやSunTagシステムを使えば、なお効果的にエピゲノム編集が可能となる。
dCas9で塩基編集
CRISPR-Cas9システムでは、ゲノム中の目的の部位にDNA二本鎖切断(DSB)を誘導し、そこにDNA修復経路を利用して変異を導入する。ところが、DSBを介さずに塩基配列を改変することもできる。これが、塩基編集である。
塩基編集のうちBEシステムでは、dCas9のN末端にシチジンデアミナーゼを融合させる。すると、sgRNAのガイドによりdCas9が結合し、この結合によって生じた一本鎖DNA部分に存在するシトシンがデアミナーゼの作用によりウラシルへと変換され、一時的にG:Uミスマッチが生じる。このようなミスマッチは通常、ウラシルDNAグリコシラーゼ(UNG)により修復されるが、dCas9のC末端に83アミノ酸のUNGの阻害因子(UGI)を融合させておくことによりこれを回避する。結果として、G:C塩基対がA:T塩基対へと変換できるのである。さらに、ニッカーゼ(nCas9)を使って脱アミノ化塩基の反対の鎖にニックを入れると、脱アミノ化塩基の正常な修復が回避され、編集効率が上昇するらしい。
同様の方法で、アデノシンデアミナーゼを使って一本鎖DNA部分のアデニンをイノシンへと変換し、A:T塩基対をG:C塩基対へと変換するABEシステムも存在する。
dCas9で特定のゲノム領域を可視化
細胞内で特定の遺伝子座を蛍光標識して観察する場合、通常は蛍光in situハイブリダイゼーション(FISH)が使われる。すなわち、細胞内でゲノムDNAを変性させ、そこに蛍光標識したプローブをハイブリダイゼーションさせる方法である。通常この方法は、固定された(死んだ)細胞のみに適用される。
生きた細胞に適用可能な方法といえば、LacOやTetOを用いたシステムがある。これは、LacオペレーターまたはTetオペレーターの反復配列をあらかじめゲノム中の目的の部位に挿入しておき、この細胞にLacリプレッサーやTetリプレッサーと蛍光タンパク質(GFPなど)の融合タンパク質を発現させる。すると、リプレッサーと蛍光タンパク質の融合タンパク質はゲノム中に挿入されたリプレッサー配列に結合し、その領域を蛍光で可視化できるのである。しかし、ゲノム中の特定の領域にこの反復配列を挿入するという大変な作業が必要であり、しかも挿入した配列がゲノムの機能に影響する可能性もある。テロメアやセントロメアのような内在の反復配列であれば、結合因子と蛍光タンパク質との融合により可視化も可能である。
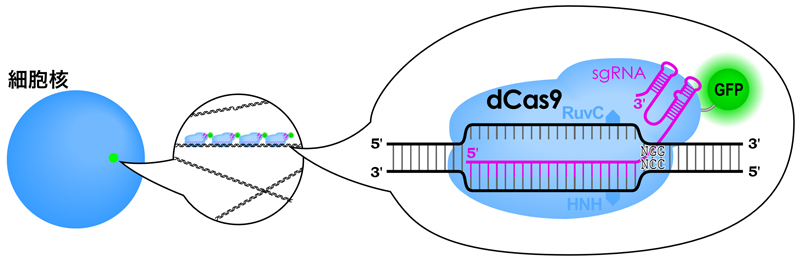
そこで登場するのが、dCas9と蛍光タンパク質の融合タンパク質である。dCas9-蛍光タンパク質を使えば、単一のsgRNAを使って生細胞中のテロメアのような反復配列を可視化できる。また、1つの遺伝子座に対する複数のsgRNAを使えば、非反復配列であっても可視化可能となる。ただしその場合、少なくとも26〜36種類のsgRNAが必要となるらしい。効率よく可視化するために、SunTagシステムを利用して1分子のdCas9に24コピーのGFPを集積させるという報告もある。また、異なるsgRNAやPAM配列を認識するdCas9(別の種に由来する)を使えば、多色での同時イメージングも可能になるのである。